+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1zf3 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | ATC Four-stranded DNA Holliday Junction | ||||||||||||||||||
 要素 要素 | 5'-D(* キーワード キーワードDNA / Crystallographic Screen / DNA Structure / Holliday Junction / Molecular Structure | 機能・相同性 | DNA |  機能・相同性情報 機能・相同性情報手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.84 Å 分子置換 / 解像度: 1.84 Å  データ登録者 データ登録者Hays, F.A. / Teegarden, A.T. / Jones, Z.J.R. / Harms, M. / Raup, D. / Watson, J. / Cavaliere, E. / Ho, P.S. |  引用 引用 ジャーナル: Proc.Natl.Acad.Sci.Usa / 年: 2005 ジャーナル: Proc.Natl.Acad.Sci.Usa / 年: 2005タイトル: How sequence defines structure: a crystallographic map of DNA structure and conformation. 著者: Hays, F.A. / Teegarden, A. / Jones, Z.J. / Harms, M. / Raup, D. / Watson, J. / Cavaliere, E. / Ho, P.S. 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1zf3.cif.gz 1zf3.cif.gz | 24.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1zf3.ent.gz pdb1zf3.ent.gz | 16.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1zf3.json.gz 1zf3.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1zf3_validation.pdf.gz 1zf3_validation.pdf.gz | 385.1 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1zf3_full_validation.pdf.gz 1zf3_full_validation.pdf.gz | 387 KB | 表示 | |
| XML形式データ |  1zf3_validation.xml.gz 1zf3_validation.xml.gz | 5.3 KB | 表示 | |
| CIF形式データ |  1zf3_validation.cif.gz 1zf3_validation.cif.gz | 7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/zf/1zf3 https://data.pdbj.org/pub/pdb/validation_reports/zf/1zf3 ftp://data.pdbj.org/pub/pdb/validation_reports/zf/1zf3 ftp://data.pdbj.org/pub/pdb/validation_reports/zf/1zf3 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  1zewC  1zexC  1zeyC  1zezC  1zf0C  1zf1C  1zf2C  1zf4C  1zf5C  1zf6C  1zf7C  1zf8C  1zf9C  1zfaC  1zfbC  1zfcC  1zfeC  1zffC  1zfgC 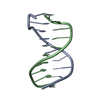 1zfhC  1zfmC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||
| 単位格子 |
| ||||||||||||
| Components on special symmetry positions |
|
- 要素
要素
| #1: DNA鎖 | 分子量: 3045.005 Da / 分子数: 2 / 由来タイプ: 合成 詳細: DNA WAS SYNTHESIZED ON AN APPLIED BIOSYSTEMS DNA SYNTHESIZER USING PHOSPHORAMIDITE CHEMISTRY, WITH THE TRITYL-PROTECTING GROUP LEFT INTACT AT THE 5'-TERMINAL NUCLEOTIDE THEN DEPROTECTED BY ...詳細: DNA WAS SYNTHESIZED ON AN APPLIED BIOSYSTEMS DNA SYNTHESIZER USING PHOSPHORAMIDITE CHEMISTRY, WITH THE TRITYL-PROTECTING GROUP LEFT INTACT AT THE 5'-TERMINAL NUCLEOTIDE THEN DEPROTECTED BY TREATMENT WITH 3% ACETIC ACID FOR FIFTEEN MINUTES, NEUTRALIZED WITH AMMONIUM HYDROXIDE, AND DESALTED ON A SIGMA G-25 SEPHADEX COLUMN. #2: 化合物 | ChemComp-CA / | #3: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.28 Å3/Da / 溶媒含有率: 46.02 % | ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 温度: 298 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 7 詳細: Na Cacodylate, CaCl2, Spermine, MPD in resevoir, pH 7.0, VAPOR DIFFUSION, SITTING DROP, temperature 298K | ||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 |
|
-データ収集
| 回折 | 平均測定温度: 103 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 14-BM-D / 波長: 0.9795 Å / ビームライン: 14-BM-D / 波長: 0.9795 Å |
| 検出器 | タイプ: ADSC QUANTUM 4 / 検出器: CCD / 日付: 2004年2月26日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.9795 Å / 相対比: 1 |
| 反射 | 解像度: 1.84→99 Å / Num. obs: 4856 / % possible obs: 98.6 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / Rmerge(I) obs: 0.061 / Rsym value: 0.061 / Χ2: 1.034 / Net I/σ(I): 15 |
| 反射 シェル | 解像度: 1.84→1.92 Å / % possible obs: 96.9 % / Rmerge(I) obs: 0.626 / Mean I/σ(I) obs: 1.07 / Num. measured obs: 465 / Rsym value: 0.626 / Χ2: 1.024 / % possible all: 96.9 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: ndb entry UD0058 解像度: 1.84→30.08 Å / Cor.coef. Fo:Fc: 0.89 / Cor.coef. Fo:Fc free: 0.851 / SU B: 6.34 / SU ML: 0.119 / TLS residual ADP flag: LIKELY RESIDUAL / 交差検証法: THROUGHOUT / ESU R: 0.212 / ESU R Free: 0.163 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 9.615 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.84→30.08 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 1.84→1.888 Å / Total num. of bins used: 20
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ | Refine-ID: X-RAY DIFFRACTION / Selection: ALL / Label seq-ID: 1 - 10
|
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj




