+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 1ykq | ||||||
|---|---|---|---|---|---|---|---|
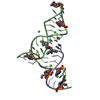
| Title | Crystal structure of Diels-Alder ribozyme | ||||||
 Components Components | (Diels-Alder ribozyme) x 2 | ||||||
 Keywords Keywords | RNA / carbon-carbon bond / catalytic mechanism / RNA crystal structure / Diels-Alder reaction / ribozyme | ||||||
| Function / homology | : / RNA / RNA (> 10) Function and homology information Function and homology information | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 3.5 Å MOLECULAR REPLACEMENT / Resolution: 3.5 Å | ||||||
 Authors Authors | Serganov, A. / Keiper, S. / Malinina, L. / Tereshko, V. / Skripkin, E. / Hobartner, C. / Polonskaia, A. / Phan, A.T. / Wombacher, R. / Micura, R. ...Serganov, A. / Keiper, S. / Malinina, L. / Tereshko, V. / Skripkin, E. / Hobartner, C. / Polonskaia, A. / Phan, A.T. / Wombacher, R. / Micura, R. / Dauter, Z. / Jaschke, A. / Patel, D.J. | ||||||
 Citation Citation |  Journal: Nat.Struct.Mol.Biol. / Year: 2005 Journal: Nat.Struct.Mol.Biol. / Year: 2005Title: Structural basis for Diels-Alder ribozyme-catalyzed carbon-carbon bond formation. Authors: Serganov, A. / Keiper, S. / Malinina, L. / Tereshko, V. / Skripkin, E. / Hobartner, C. / Polonskaia, A. / Phan, A.T. / Wombacher, R. / Micura, R. / Dauter, Z. / Jaschke, A. / Patel, D.J. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  1ykq.cif.gz 1ykq.cif.gz | 57.9 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb1ykq.ent.gz pdb1ykq.ent.gz | 40.8 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  1ykq.json.gz 1ykq.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/yk/1ykq https://data.pdbj.org/pub/pdb/validation_reports/yk/1ykq ftp://data.pdbj.org/pub/pdb/validation_reports/yk/1ykq ftp://data.pdbj.org/pub/pdb/validation_reports/yk/1ykq | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  1ykvC  1ylsSC C: citing same article ( S: Starting model for refinement |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||
| Unit cell |
| ||||||||||||||||||||||||||||||||||||||||||||||||
| Noncrystallographic symmetry (NCS) | NCS domain:
NCS domain segments: Component-ID: 1 / Beg auth comp-ID: G / Beg label comp-ID: G / End auth comp-ID: C / End label comp-ID: C / Refine code: 6
NCS ensembles :
|
- Components
Components
| #1: RNA chain | Mass: 3497.146 Da / Num. of mol.: 2 / Source method: obtained synthetically Details: This sequence was derived from the in vitro selected ribozyme catalyzing Diels-Alder reaction between tethered anthracene and biotinylated maleimide #2: RNA chain | Mass: 12262.333 Da / Num. of mol.: 2 / Source method: obtained synthetically Details: This sequence was derived from the in vitro selected ribozyme catalyzing Diels-Alder reaction between tethered anthracene and biotinylated maleimide #3: Chemical | ChemComp-CD / #4: Chemical | ChemComp-MG / |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 4.04 Å3/Da / Density % sol: 63 % | ||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Crystal grow | Temperature: 293 K / Method: vapor diffusion, hanging drop / pH: 6.5 Details: PEG400, sodium cacodylate, magnesium acetate, 1,6-hexandiol, sodium chloride, cadmium chloride, pH 6.5, VAPOR DIFFUSION, HANGING DROP, temperature 293K | ||||||||||||||||||||||||||||||||||||||||||||
| Components of the solutions |
|
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  NSLS NSLS  / Beamline: X25 / Wavelength: 1.1 Å / Beamline: X25 / Wavelength: 1.1 Å |
| Detector | Type: ADSC QUANTUM 315 / Detector: CCD / Date: Jun 13, 2004 |
| Radiation | Monochromator: Si(111),(220) / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1.1 Å / Relative weight: 1 |
| Reflection | Resolution: 3.5→20 Å / Num. all: 6902 / Num. obs: 6744 / % possible obs: 97.7 % / Observed criterion σ(I): 0 / Redundancy: 3.8 % / Rmerge(I) obs: 0.154 / Net I/σ(I): 8.1 |
| Reflection shell | Resolution: 3.5→3.62 Å / Redundancy: 3.8 % / Rmerge(I) obs: 0.729 / Mean I/σ(I) obs: 1.8 / Num. unique all: 673 / % possible all: 98.2 |
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: Diels-Alder ribozyme from the ribozyme-product complex, pdb entry 1YLS Resolution: 3.5→20 Å / Cor.coef. Fo:Fc: 0.896 / Cor.coef. Fo:Fc free: 0.852 / SU B: 109.285 / SU ML: 0.73 / TLS residual ADP flag: LIKELY RESIDUAL / Isotropic thermal model: overall / Cross valid method: THROUGHOUT / σ(F): 0 / ESU R Free: 0.671 / Stereochemistry target values: MAXIMUM LIKELIHOOD
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Ion probe radii: 0.8 Å / Shrinkage radii: 0.8 Å / VDW probe radii: 1.2 Å / Solvent model: BABINET MODEL WITH MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 40 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.755 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 3.5→20 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints NCS | Dom-ID: 1 / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Resolution: 3.5→3.588 Å / Total num. of bins used: 20 /
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller










 PDBj
PDBj

































