| 登録情報 | データベース: PDB / ID: 1xnk
|
|---|
| タイトル | Beta-1,4-xylanase from Chaetomium thermophilum complexed with methyl thioxylopentoside |
|---|
 要素 要素 | endoxylanase 11A |
|---|
 キーワード キーワード | HYDROLASE / xylanase / glycoside hydrolase / family 11 / glycosidase / thioinhibitor / sulphur containing inhibitor |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
endo-1,4-beta-xylanase activity / endo-1,4-beta-xylanase / xylan catabolic process類似検索 - 分子機能 Glycoside hydrolase family 11/12, catalytic domain / Glycoside hydrolase family 11, active site 1 / Glycosyl hydrolases family 11 (GH11) active site signature 1. / Glycoside hydrolase family 11 / Glycosyl hydrolases family 11 (GH11) domain / Glycosyl hydrolases family 11 / Glycosyl hydrolases family 11 (GH11) domain profile. / Glycoside hydrolase family 11/12 / Concanavalin A-like lectin/glucanase domain superfamily / Jelly Rolls ...Glycoside hydrolase family 11/12, catalytic domain / Glycoside hydrolase family 11, active site 1 / Glycosyl hydrolases family 11 (GH11) active site signature 1. / Glycoside hydrolase family 11 / Glycosyl hydrolases family 11 (GH11) domain / Glycosyl hydrolases family 11 / Glycosyl hydrolases family 11 (GH11) domain profile. / Glycoside hydrolase family 11/12 / Concanavalin A-like lectin/glucanase domain superfamily / Jelly Rolls / Sandwich / Mainly Beta類似検索 - ドメイン・相同性 methyl pentopyranosyl-(1->4)-4-thiopentopyranosyl-(1->4)-4-thio-beta-D-xylopyranosyl-(1->4)-4-thio-beta-D-xylopyranosyl-(1->4)- 4-thio-alpha-D-xylopyranoside / Endo-1,4-beta-xylanase類似検索 - 構成要素 |
|---|
| 生物種 |  Chaetomium thermophilum (菌類) Chaetomium thermophilum (菌類) |
|---|
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 1.55 Å 分子置換 / 解像度: 1.55 Å |
|---|
 データ登録者 データ登録者 | Hakanpaa, J. / Hakulinen, N. / Rouvinen, J. |
|---|
 引用 引用 |  ジャーナル: FEBS J. / 年: 2005 ジャーナル: FEBS J. / 年: 2005
タイトル: Determination of thioxylo-oligosaccharide binding to family 11 xylanases using electrospray ionization Fourier transform ion cyclotron resonance mass spectrometry and X-ray crystallography
著者: Janis, J. / Hakanpaa, J. / Hakulinen, N. / Ibatullin, F.M. / Hoxha, A. / Derrick, P.J. / Rouvinen, J. / Vainiotalo, P. |
|---|
| 履歴 | | 登録 | 2004年10月5日 | 登録サイト: RCSB / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2005年5月10日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年4月30日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Derived calculations / Version format compliance |
|---|
| 改定 1.3 | 2017年10月11日 | Group: Refinement description / カテゴリ: software |
|---|
| 改定 1.4 | 2018年7月11日 | Group: Data collection / カテゴリ: diffrn_source / Item: _diffrn_source.type |
|---|
| 改定 2.0 | 2019年12月25日 | Group: Database references / Derived calculations / Polymer sequence
カテゴリ: entity_poly / pdbx_struct_mod_residue ...entity_poly / pdbx_struct_mod_residue / struct_conn / struct_ref_seq_dif
Item: _entity_poly.pdbx_seq_one_letter_code_can / _pdbx_struct_mod_residue.parent_comp_id / _struct_conn.pdbx_leaving_atom_flag |
|---|
| 改定 3.0 | 2020年7月29日 | Group: Advisory / Atomic model ...Advisory / Atomic model / Data collection / Derived calculations / Non-polymer description / Structure summary
カテゴリ: atom_site / chem_comp ...atom_site / chem_comp / entity / pdbx_branch_scheme / pdbx_entity_branch / pdbx_entity_branch_descriptor / pdbx_entity_branch_link / pdbx_entity_branch_list / pdbx_entity_nonpoly / pdbx_nonpoly_scheme / pdbx_struct_assembly_gen / pdbx_struct_special_symmetry / pdbx_unobs_or_zero_occ_atoms / struct_asym / struct_conn / struct_site / struct_site_gen
Item: _atom_site.B_iso_or_equiv / _atom_site.Cartn_x ..._atom_site.B_iso_or_equiv / _atom_site.Cartn_x / _atom_site.Cartn_y / _atom_site.Cartn_z / _atom_site.auth_asym_id / _atom_site.auth_atom_id / _atom_site.auth_comp_id / _atom_site.auth_seq_id / _atom_site.label_asym_id / _atom_site.label_atom_id / _atom_site.label_comp_id / _atom_site.label_entity_id / _atom_site.occupancy / _atom_site.type_symbol / _chem_comp.formula / _chem_comp.formula_weight / _chem_comp.id / _chem_comp.mon_nstd_flag / _chem_comp.name / _chem_comp.type / _entity.formula_weight / _entity.pdbx_description / _entity.pdbx_number_of_molecules / _entity.src_method / _entity.type / _pdbx_struct_assembly_gen.asym_id_list / _pdbx_struct_special_symmetry.label_asym_id / _struct_asym.entity_id
解説: Carbohydrate remediation / Provider: repository / タイプ: Remediation |
|---|
| 改定 3.1 | 2020年10月14日 | Group: Structure summary / カテゴリ: pdbx_molecule_features |
|---|
| 改定 3.2 | 2023年10月25日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
| 改定 3.3 | 2024年10月23日 | Group: Structure summary
カテゴリ: pdbx_entry_details / pdbx_modification_feature |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Chaetomium thermophilum (菌類)
Chaetomium thermophilum (菌類) X線回折 /
X線回折 /  分子置換 / 解像度: 1.55 Å
分子置換 / 解像度: 1.55 Å  データ登録者
データ登録者 引用
引用 ジャーナル: FEBS J. / 年: 2005
ジャーナル: FEBS J. / 年: 2005 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1xnk.cif.gz
1xnk.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1xnk.ent.gz
pdb1xnk.ent.gz PDB形式
PDB形式 1xnk.json.gz
1xnk.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 1xnk_validation.pdf.gz
1xnk_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 1xnk_full_validation.pdf.gz
1xnk_full_validation.pdf.gz 1xnk_validation.xml.gz
1xnk_validation.xml.gz 1xnk_validation.cif.gz
1xnk_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/xn/1xnk
https://data.pdbj.org/pub/pdb/validation_reports/xn/1xnk ftp://data.pdbj.org/pub/pdb/validation_reports/xn/1xnk
ftp://data.pdbj.org/pub/pdb/validation_reports/xn/1xnk
 リンク
リンク 集合体
集合体



 要素
要素 Chaetomium thermophilum (菌類) / 発現宿主:
Chaetomium thermophilum (菌類) / 発現宿主:  Hypocrea jecorina (菌類) / 参照: UniProt: Q8J1V6, endo-1,4-beta-xylanase
Hypocrea jecorina (菌類) / 参照: UniProt: Q8J1V6, endo-1,4-beta-xylanase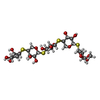
 タイプ: oligosaccharide, Oligosaccharide / クラス: 基質類似体 / 分子量: 476.582 Da / 分子数: 2 / 由来タイプ: 組換発現
タイプ: oligosaccharide, Oligosaccharide / クラス: 基質類似体 / 分子量: 476.582 Da / 分子数: 2 / 由来タイプ: 組換発現 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 回転陽極 / タイプ: RIGAKU RU200 / 波長: 1.5418 Å
回転陽極 / タイプ: RIGAKU RU200 / 波長: 1.5418 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj



