+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 1ugi | ||||||
|---|---|---|---|---|---|---|---|
| Title | URACIL-DNA GLYCOSYLASE INHIBITOR PROTEIN | ||||||
 Components Components | URACIL-DNA GLYCOSYLASE INHIBITOR | ||||||
 Keywords Keywords | HYDROLASE INHIBITOR / PROTEIN MIMICRY OF DNA / PROTEIN INHIBITOR | ||||||
| Function / homology | Bacteriophage PBS2, uracil-glycosylase inhibitor / Bacteriophage PBS2, uracil-glycosylase inhibitor / Uracil-DNA glycosylase inhibitor / Uracil-DNA glycosylase inhibitor / Nuclear Transport Factor 2; Chain: A, / Roll / Alpha Beta / IMIDAZOLE / Uracil-DNA glycosylase inhibitor Function and homology information Function and homology information | ||||||
| Biological species |  Bacillus phage PBS2 (virus) Bacillus phage PBS2 (virus) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.55 Å MOLECULAR REPLACEMENT / Resolution: 1.55 Å | ||||||
 Authors Authors | Putnam, C.D. / Arvai, A.S. / Mol, C.D. / Tainer, J.A. | ||||||
 Citation Citation |  Journal: J.Mol.Biol. / Year: 1999 Journal: J.Mol.Biol. / Year: 1999Title: Protein mimicry of DNA from crystal structures of the uracil-DNA glycosylase inhibitor protein and its complex with Escherichia coli uracil-DNA glycosylase Authors: Putnam, C.D. / Shroyer, M.J. / Lundquist, A.J. / Mol, C.D. / Arvai, A.S. / Mosbaugh, D.W. / Tainer, J.A. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  1ugi.cif.gz 1ugi.cif.gz | 157 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb1ugi.ent.gz pdb1ugi.ent.gz | 126.3 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  1ugi.json.gz 1ugi.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/ug/1ugi https://data.pdbj.org/pub/pdb/validation_reports/ug/1ugi ftp://data.pdbj.org/pub/pdb/validation_reports/ug/1ugi ftp://data.pdbj.org/pub/pdb/validation_reports/ug/1ugi | HTTPS FTP |
|---|
-Related structure data
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||||||||||||||||||||||
| 2 | 
| ||||||||||||||||||||||||||||||||
| 3 | 
| ||||||||||||||||||||||||||||||||
| 4 | 
| ||||||||||||||||||||||||||||||||
| 5 | 
| ||||||||||||||||||||||||||||||||
| 6 | 
| ||||||||||||||||||||||||||||||||
| 7 | 
| ||||||||||||||||||||||||||||||||
| 8 | 
| ||||||||||||||||||||||||||||||||
| Unit cell |
| ||||||||||||||||||||||||||||||||
| Noncrystallographic symmetry (NCS) | NCS oper:
|
- Components
Components
| #1: Protein | Mass: 9482.674 Da / Num. of mol.: 8 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Bacillus phage PBS2 (virus) / Cellular location: CYTOPLASM / Plasmid: PZWTAC1 / Cellular location (production host): CYTOPLASM / Gene (production host): TAC / Production host: Bacillus phage PBS2 (virus) / Cellular location: CYTOPLASM / Plasmid: PZWTAC1 / Cellular location (production host): CYTOPLASM / Gene (production host): TAC / Production host:  #2: Chemical | ChemComp-SO4 / #3: Chemical | ChemComp-IMD / #4: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.6 Å3/Da / Density % sol: 53 % | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Crystal grow | Temperature: 277 K / pH: 8.2 / Details: pH 8.2, temperature 277K | ||||||||||||||||||||
| Components of the solutions |
| ||||||||||||||||||||
| Crystal grow | *PLUS Method: vapor diffusion | ||||||||||||||||||||
| Components of the solutions | *PLUS
|
-Data collection
| Diffraction | Mean temperature: 150 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  SSRL SSRL  / Beamline: BL9-1 / Wavelength: 0.918 / Beamline: BL9-1 / Wavelength: 0.918 |
| Detector | Type: MARRESEARCH / Detector: IMAGE PLATE |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.918 Å / Relative weight: 1 |
| Reflection | Resolution: 1.55→30 Å / Num. obs: 95452 / % possible obs: 86.7 % / Redundancy: 2.3 % / Rsym value: 0.064 / Net I/σ(I): 16.9 |
| Reflection shell | Resolution: 1.55→1.59 Å / Redundancy: 2.3 % / Mean I/σ(I) obs: 2 / Rsym value: 0.368 / % possible all: 86.5 |
| Reflection | *PLUS Num. measured all: 221922 / Rmerge(I) obs: 0.064 |
| Reflection shell | *PLUS % possible obs: 86.5 % / Rmerge(I) obs: 0.368 / Mean I/σ(I) obs: 2 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: ORTHORHOMBIC CRYSTAL FORM OF FREE UGI Resolution: 1.55→20 Å / Num. parameters: 24407 / Num. restraintsaints: 22880 / Cross valid method: THROUGHOUT / σ(F): 0 / Stereochemistry target values: ENGH AND HUBER
| |||||||||||||||||||||||||||||||||
| Solvent computation | Solvent model: BABINET'S PRINCIPLE SCALING | |||||||||||||||||||||||||||||||||
| Refine analyze | Num. disordered residues: 24 | |||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 1.55→20 Å
| |||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||
| Software | *PLUS Name: SHELXL-97 / Classification: refinement | |||||||||||||||||||||||||||||||||
| Refinement | *PLUS Rfactor obs: 0.222 / Rfactor Rfree: 0.289 | |||||||||||||||||||||||||||||||||
| Solvent computation | *PLUS | |||||||||||||||||||||||||||||||||
| Displacement parameters | *PLUS | |||||||||||||||||||||||||||||||||
| LS refinement shell | *PLUS Highest resolution: 1.55 Å / Lowest resolution: 1.62 Å / Rfactor Rfree: 0.348 / Num. reflection obs: 9225 / Rfactor obs: 0.329 |
 Movie
Movie Controller
Controller













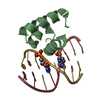

 PDBj
PDBj









