+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1u2u | ||||||
|---|---|---|---|---|---|---|---|
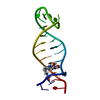
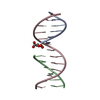
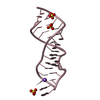
| タイトル | Nmr solution structure of a designed heterodimeric leucine zipper | ||||||
 要素 要素 | (General control protein GCN4) x 2 | ||||||
 キーワード キーワード | TRANSCRIPTION / COILED COIL / LEUCINE ZIPPER / INTER-HELICAL ION PAIRING / ELECTROSTATIC INTERACTIONS | ||||||
| 手法 | 溶液NMR / DISTANCE GEOMETRY, REGULARIZATION, SIMULATED ANNEALING, MOLECULAR DYNAMICS SIMULATION IN VACUO | ||||||
 データ登録者 データ登録者 | Marti, D.N. / Bosshard, H.R. | ||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 2004 ジャーナル: Biochemistry / 年: 2004タイトル: Inverse electrostatic effect: electrostatic repulsion in the unfolded state stabilizes a leucine zipper. 著者: Marti, D.N. / Bosshard, H.R. #1: ジャーナル: J.Mol.Biol. / 年: 2003 タイトル: Electrostatic interactions in leucine zippers: thermodynamic analysis of the contributions of Glu and His residues and the effect of mutating salt bridges 著者: Marti, D.N. / Bosshard, H.R. #2:  ジャーナル: Biochemistry / 年: 2000 ジャーナル: Biochemistry / 年: 2000タイトル: Interhelical ion pairing in coiled coils: solution structure of a heterodimeric leucine zipper and determination of pKa values of Glu side chains 著者: Marti, D.N. / Jelesarov, I. / Bosshard, H.R. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1u2u.cif.gz 1u2u.cif.gz | 517.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1u2u.ent.gz pdb1u2u.ent.gz | 437 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1u2u.json.gz 1u2u.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1u2u_validation.pdf.gz 1u2u_validation.pdf.gz | 359.2 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1u2u_full_validation.pdf.gz 1u2u_full_validation.pdf.gz | 679.2 KB | 表示 | |
| XML形式データ |  1u2u_validation.xml.gz 1u2u_validation.xml.gz | 32.9 KB | 表示 | |
| CIF形式データ |  1u2u_validation.cif.gz 1u2u_validation.cif.gz | 52.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/u2/1u2u https://data.pdbj.org/pub/pdb/validation_reports/u2/1u2u ftp://data.pdbj.org/pub/pdb/validation_reports/u2/1u2u ftp://data.pdbj.org/pub/pdb/validation_reports/u2/1u2u | HTTPS FTP |
-関連構造データ
| 関連構造データ | |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質・ペプチド | 分子量: 3480.719 Da / 分子数: 1 / 由来タイプ: 合成 詳細: Synthetic peptide based on the sequence of the leucine zipper domain in GCN4 (baker's yeast) |
|---|---|
| #2: タンパク質・ペプチド | 分子量: 3508.238 Da / 分子数: 1 / 由来タイプ: 合成 詳細: synthetic peptide based on the sequence of the leucine zipper domain in GCN4 (baker's yeast) |
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
| ||||||||||||||||||||||||
| NMR実験の詳細 | Text: STRUCTURE WAS DETERMINED USING STANDARD 2D NMR TECHNIQUES |
- 試料調製
試料調製
| 詳細 | 内容: 2.6mM, 90% H2O, 10% D2O / 溶媒系: 90% H2O/10% D2O |
|---|---|
| 試料状態 | イオン強度: 10mM / pH: 5.7 / 圧: AMBIENT / 温度: 310.00 K |
-NMR測定
| NMRスペクトロメーター | タイプ: Bruker DRX / 製造業者: Bruker / モデル: DRX / 磁場強度: 600 MHz |
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: DISTANCE GEOMETRY, REGULARIZATION, SIMULATED ANNEALING, MOLECULAR DYNAMICS SIMULATION IN VACUO ソフトェア番号: 1 詳細: STRUCTURES WERE CALCULATED ON THE BASIS OF 1246 NOE DERIVED DISTANCE CONSTRAINTS AND 44 PHI ANGLE RESTRAINTS | ||||||||||||||||||||||||||||
| 代表構造 | 選択基準: closest to the average | ||||||||||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: structures with the least restraint violations, structures with the lowest energy, low deviation of experimental pKa values from pKa derived by continuum ...コンフォーマー選択の基準: structures with the least restraint violations, structures with the lowest energy, low deviation of experimental pKa values from pKa derived by continuum electrostatics calculations on structures 計算したコンフォーマーの数: 50 / 登録したコンフォーマーの数: 27 |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj HSQC
HSQC