| 登録情報 | データベース: PDB / ID: 1tou
|
|---|
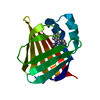
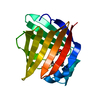
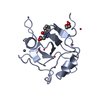
| タイトル | Crystal structure of human adipocyte fatty acid binding protein in complex with a non-covalent ligand |
|---|
 要素 要素 | Fatty acid-binding protein, adipocyte |
|---|
 キーワード キーワード | LIPID TRANSPORT / Transport / Lipid-binding |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
hormone receptor binding / long-chain fatty acid transmembrane transporter activity / long-chain fatty acid binding / cellular response to lithium ion / Triglyceride catabolism / white fat cell differentiation / long-chain fatty acid transport / fatty acid transport / brown fat cell differentiation / lipid droplet ...hormone receptor binding / long-chain fatty acid transmembrane transporter activity / long-chain fatty acid binding / cellular response to lithium ion / Triglyceride catabolism / white fat cell differentiation / long-chain fatty acid transport / fatty acid transport / brown fat cell differentiation / lipid droplet / cholesterol homeostasis / fatty acid binding / response to bacterium / Transcriptional regulation of white adipocyte differentiation / positive regulation of inflammatory response / cellular response to tumor necrosis factor / positive regulation of cold-induced thermogenesis / MLL4 and MLL3 complexes regulate expression of PPARG target genes in adipogenesis and hepatic steatosis / negative regulation of DNA-templated transcription / extracellular exosome / nucleus / cytosol / cytoplasm類似検索 - 分子機能 Cytosolic fatty-acid binding proteins signature. / Intracellular lipid binding protein / Cytosolic fatty-acid binding / Lipocalin / cytosolic fatty-acid binding protein family / Lipocalin/cytosolic fatty-acid binding domain / Calycin beta-barrel core domain / Calycin / Lipocalin / Beta Barrel / Mainly Beta類似検索 - ドメイン・相同性 Chem-B1V / Fatty acid-binding protein, adipocyte類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  フーリエ合成 / 解像度: 2 Å フーリエ合成 / 解像度: 2 Å |
|---|
 データ登録者 データ登録者 | Ringom, R. / Axen, E. / Uppenberg, J. / Lundback, T. / Rondahl, L. / Barf, T. |
|---|
 引用 引用 |  ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2004 ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2004
タイトル: Substituted benzylamino-6-(trifluoromethyl)pyrimidin-4(1H)-ones: a novel class of selective human A-FABP inhibitors.
著者: Ringom, R. / Axen, E. / Uppenberg, J. / Lundback, T. / Rondahl, L. / Barf, T. |
|---|
| 履歴 | | 登録 | 2004年6月15日 | 登録サイト: RCSB / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2004年8月24日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年4月30日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2024年3月13日 | Group: Data collection / Database references / Derived calculations
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  フーリエ合成 / 解像度: 2 Å
フーリエ合成 / 解像度: 2 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2004
ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2004 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1tou.cif.gz
1tou.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1tou.ent.gz
pdb1tou.ent.gz PDB形式
PDB形式 1tou.json.gz
1tou.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/to/1tou
https://data.pdbj.org/pub/pdb/validation_reports/to/1tou ftp://data.pdbj.org/pub/pdb/validation_reports/to/1tou
ftp://data.pdbj.org/pub/pdb/validation_reports/to/1tou リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 発現宿主:
Homo sapiens (ヒト) / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 回転陽極 / タイプ: RIGAKU / 波長: 1.5418 Å
回転陽極 / タイプ: RIGAKU / 波長: 1.5418 Å 解析
解析 フーリエ合成 / 解像度: 2→15 Å / Cor.coef. Fo:Fc: 0.939 / Cor.coef. Fo:Fc free: 0.889 / SU B: 5.057 / SU ML: 0.142 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.245 / ESU R Free: 0.219 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD
フーリエ合成 / 解像度: 2→15 Å / Cor.coef. Fo:Fc: 0.939 / Cor.coef. Fo:Fc free: 0.889 / SU B: 5.057 / SU ML: 0.142 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.245 / ESU R Free: 0.219 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj




