+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1rl4 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Plasmodium falciparum peptide deformylase complex with inhibitor | ||||||
 要素 要素 | formylmethionine deformylase | ||||||
 キーワード キーワード | HYDROLASE / crystal engineering / drug design / malaria / PDF / peptide deformylase / Plasmodium | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報N-terminal protein amino acid modification / apicoplast / peptide deformylase / peptide deformylase activity / : / ferrous iron binding / translation / membrane 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.18 Å 分子置換 / 解像度: 2.18 Å | ||||||
 データ登録者 データ登録者 | Robien, M.A. / Nguyen, K.T. / Kumar, A. / Hirsh, I. / Turley, S. / Pei, D. / Hol, W.G.J. | ||||||
 引用 引用 |  ジャーナル: Protein Sci. / 年: 2004 ジャーナル: Protein Sci. / 年: 2004タイトル: An improved crystal form of Plasmodium falciparum peptide deformylase. 著者: Robien, M.A. / Nguyen, K.T. / Kumar, A. / Hirsh, I. / Turley, S. / Pei, D. / Hol, W.G.J. #1:  ジャーナル: Structure / 年: 2002 ジャーナル: Structure / 年: 2002タイトル: Crystals of Peptide Deformylase from Plasmodium falciparum Reveal Critical Characteristics of the Active Site for Drug Design 著者: Kumar, A. / Nguyen, K.T. / Srivathsan, S. / Ornstein, B. / Turley, S. / Hirsh, I. / Pei, D. / Hol, W.G.J. | ||||||
| 履歴 |
| ||||||
| Remark 600 | HETEROGEN AS DISCUSSED IN REFERENCE 1 (ROBIEN ET AL, PROTEIN SCIENCE), THE MODEL CHOSEN FOR BL5, ...HETEROGEN AS DISCUSSED IN REFERENCE 1 (ROBIEN ET AL, PROTEIN SCIENCE), THE MODEL CHOSEN FOR BL5, PARTICULARLY IN THE ATOMS LINKING THE TWO HALVES OF THIS MOLECULE, WAS CHOSEN BASED ON THE ELECTRON DENSITY FEATURE USING OMIT MAPS. FROM THE PROTEIN SCIENCE MANUSCRIPT: ALTHOUGH THE IDENTITY OF THE LINKER ATOMS ARE UNKNOWN, A LINEAR -CH2-NH-NH-CH2- CHAIN IS APPROXIMATELY THE CORRECT SIZE. BECAUSE OF THE UNCERTAINTY PERTAINING TO THE LINKER, THE MODEL CHOSEN FOR THE LINKER SHOULD BE CONSIDERED AS EXPLORATORY AND PROVISIONAL, RATHER THAN CONCLUSIVE. WATERS 701-753 AND 801-818, CO 301, BRR 401, AND BL5 501 ARE ASSOCIATED WITH CHAIN A. WATERS 1701-1753 AND 901-953, CO 1301, BRR 1401, AND BL5 1501 ARE ASSOCIATED WITH CHAIN B. | ||||||
| Remark 999 | SEQUENCE THE NUMBERING OF RESIDUES IS BASED ON THE FULL SEQUENCE OF THE P.FALCIPARUM PDF PROTEIN, ...SEQUENCE THE NUMBERING OF RESIDUES IS BASED ON THE FULL SEQUENCE OF THE P.FALCIPARUM PDF PROTEIN, WHICH HAS A LARGE N-TERMINAL (APICOPLAST TARGETING) SIGNAL SEQUENCE, WHICH IS TRUNCATED IN VIVO IN THE PROCESS OF TRANSPORT INTO THE APICOPLAST ORGANELLE. A CONSTRUCT WITH AN N-TERMINAL 57 RESIDUE TRUNCATION WAS EXPRESSED IN E COLI FOR THIS STUDY. THE C-TERMINAL SEQUENCE USED IN THIS STUDY DIFFERS FROM THAT USED FOR PDB ENTRY 1JYM BY THE DELETION OF THREE RESIDUES (-EEP-) JUST PRIOR TO THE -LEHHHHHH OF THE HEXAHISTADINE TAG. THIS MODIFICATION WAS UNDERTAKEN AS A SUCCESSFUL EFFORT OF CRYSTAL ENGINEERING TO AVOID A NETWORK OF UNDESIRABLE CRYSTAL CONTACTS IN THE PREVIOUS STUDY. THE SEQUENCE OF P. FALCIPARUM PDF IN THIS DEPOSITION INCLUDES TWO SINGLE AMINO ACID CORRECTIONS TO THE SEQUENCE REPORTED IN THE ORIGINAL DEPOSITION (1JYM) OF P. FALCIPARUM PDF. RESIDUES GLU81 AND PRO160 HAD BEEN INADVERTENTLY NOT INCLUDED IN THE SEQUENCE USED DURING THE TRACING OF THE ELECTRON DENSITY OF THE ORIGINAL STUDY. THE RESIDUE NUMBERING OF THIS DEPOSITION REFLECTS THE MODIFIED, CORRECTED SEQUENCE INFORMATION, AND NOW AGREES WITH THE SEQUENCE DATA DERIVED FROM THE DEPOSITED P. FALCIPARUM GENOME. |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1rl4.cif.gz 1rl4.cif.gz | 89.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1rl4.ent.gz pdb1rl4.ent.gz | 65.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1rl4.json.gz 1rl4.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/rl/1rl4 https://data.pdbj.org/pub/pdb/validation_reports/rl/1rl4 ftp://data.pdbj.org/pub/pdb/validation_reports/rl/1rl4 ftp://data.pdbj.org/pub/pdb/validation_reports/rl/1rl4 | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | 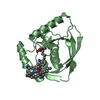
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 単位格子 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域:
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj