+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1rh3 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | DIHYDROFOLATE REDUCTASE COMPLEXED WITH METHOTREXATE AND NICOTINAMIDE ADENINE DINUCLEOTIDE PHOSPHATE (REDUCED FORM) | ||||||
 要素 要素 | DIHYDROFOLATE REDUCTASE | ||||||
 キーワード キーワード | OXIDOREDUCTASE / NADP / TRIMETHOPRIM RESISTANCE / METHOTREXATE RESISTANCE / ONE-CARBON METABOLISM | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報methotrexate binding / dihydrofolic acid binding / 10-formyltetrahydrofolate biosynthetic process / response to methotrexate / folic acid biosynthetic process / folic acid binding / NADP+ binding / dihydrofolate metabolic process / dihydrofolate reductase / dihydrofolate reductase activity ...methotrexate binding / dihydrofolic acid binding / 10-formyltetrahydrofolate biosynthetic process / response to methotrexate / folic acid biosynthetic process / folic acid binding / NADP+ binding / dihydrofolate metabolic process / dihydrofolate reductase / dihydrofolate reductase activity / folic acid metabolic process / NADPH binding / tetrahydrofolate biosynthetic process / one-carbon metabolic process / NADP binding / response to xenobiotic stimulus / response to antibiotic / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / DIFFERENCE FOURIER / 解像度: 2.4 Å X線回折 / DIFFERENCE FOURIER / 解像度: 2.4 Å | ||||||
 データ登録者 データ登録者 | Sawaya, M.R. / Kraut, J. | ||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 1997 ジャーナル: Biochemistry / 年: 1997タイトル: Loop and subdomain movements in the mechanism of Escherichia coli dihydrofolate reductase: crystallographic evidence. 著者: Sawaya, M.R. / Kraut, J. #1:  ジャーナル: Biochemistry / 年: 1995 ジャーナル: Biochemistry / 年: 1995タイトル: Isomorphous Crystal Structures of Escherichia Coli Dihydrofolate Reductase Complexed with Folate, 5-Deazafolate, and 5,10-Dideazatetrahydrofolate: Mechanistic Implications 著者: Reyes, V.M. / Sawaya, M.R. / Brown, K.A. / Kraut, J. #2:  ジャーナル: Biochemistry / 年: 1991 ジャーナル: Biochemistry / 年: 1991タイトル: Crystal Structure of Unliganded Escherichia Coli Dihydrofolate Reductase. Ligand-Induced Conformational Changes and Cooperativity in Binding 著者: Bystroff, C. / Kraut, J. #3:  ジャーナル: Biochemistry / 年: 1990 ジャーナル: Biochemistry / 年: 1990タイトル: Crystal Structures of Escherichia Coli Dihydrofolate Reductase: The Nadp+ Holoenzyme and the Folate.Nadp+ Ternary Complex. Substrate Binding and a Model for the Transition State 著者: Bystroff, C. / Oatley, S.J. / Kraut, J. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1rh3.cif.gz 1rh3.cif.gz | 48.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1rh3.ent.gz pdb1rh3.ent.gz | 33.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1rh3.json.gz 1rh3.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1rh3_validation.pdf.gz 1rh3_validation.pdf.gz | 1017.3 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1rh3_full_validation.pdf.gz 1rh3_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  1rh3_validation.xml.gz 1rh3_validation.xml.gz | 13.7 KB | 表示 | |
| CIF形式データ |  1rh3_validation.cif.gz 1rh3_validation.cif.gz | 16.4 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/rh/1rh3 https://data.pdbj.org/pub/pdb/validation_reports/rh/1rh3 ftp://data.pdbj.org/pub/pdb/validation_reports/rh/1rh3 ftp://data.pdbj.org/pub/pdb/validation_reports/rh/1rh3 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  1dreC  1ra1C  1ra2C  1ra3C  1ra8C  1ra9C  1rb2C  1rb3C  1rc4C  1rd7C  1re7C  1rf7C  1rg7C  1rx1C  1rx2C  1rx3C  1rx4C  1rx5C  1rx6C  1rx7C  1rx8C  1rx9C  1rh2S S: 精密化の開始モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 18020.326 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   |
|---|---|
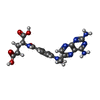
| #2: 化合物 | ChemComp-MTX / |
| #3: 化合物 | ChemComp-NAP / |
| #4: 水 | ChemComp-HOH / |
| Has protein modification | N |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.31 Å3/Da / 溶媒含有率: 62.9 % | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | pH: 7 / 詳細: pH 7.0 | ||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 手法: macro seeding | ||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 298 K |
|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: RIGAKU RUH2R / 波長: 1.5418 回転陽極 / タイプ: RIGAKU RUH2R / 波長: 1.5418 |
| 検出器 | タイプ: XUONG-HAMLIN MULTIWIRE / 検出器: AREA DETECTOR / 日付: 1991年8月8日 |
| 放射 | モノクロメーター: GRAPHITE(002) / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.5418 Å / 相対比: 1 |
| 反射 | 解像度: 2.4→100 Å / Num. obs: 9577 / % possible obs: 94 % / Observed criterion σ(I): 0 / 冗長度: 3 % / Rsym value: 0.057 / Net I/σ(I): 17.8 |
| 反射 シェル | 解像度: 2.4→2.6 Å / 冗長度: 2.1 % / Mean I/σ(I) obs: 2.5 / Rsym value: 0.218 / % possible all: 87 |
| 反射 | *PLUS Num. measured all: 36699 / Rmerge(I) obs: 0.057 |
| 反射 シェル | *PLUS % possible obs: 87 % / Rmerge(I) obs: 0.218 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法: DIFFERENCE FOURIER 開始モデル: PDB ENTRY 1RH2 解像度: 2.4→20 Å / σ(F): 0 / 立体化学のターゲット値: TNT PROTGEO /
| ||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: MOEWS AND KRETSINGER / Bsol: 321.7 Å2 / ksol: 0.871 e/Å3 | ||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.4→20 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: TNT / バージョン: 5-D / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS Rfactor obs: 0.228 | ||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS タイプ: t_plane_restr / Dev ideal: 0.006 / Weight: 0.02 |
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj