[English] 日本語
 Yorodumi
Yorodumi- PDB-1qxd: Structural Basis for the Potent Antisickling Effect of a Novel Cl... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 1qxd | ||||||
|---|---|---|---|---|---|---|---|
| Title | Structural Basis for the Potent Antisickling Effect of a Novel Class of 5-Membered Heterocyclic Aldehydic Compounds | ||||||
 Components Components |
| ||||||
 Keywords Keywords | OXYGEN STORAGE/TRANSPORT / allosteric / antisickling / relaxed state / hemoglobin / sickle cell / furfural / OXYGEN STORAGE-TRANSPORT COMPLEX | ||||||
| Function / homology |  Function and homology information Function and homology informationnitric oxide transport / hemoglobin alpha binding / cellular oxidant detoxification / hemoglobin binding / haptoglobin-hemoglobin complex / renal absorption / hemoglobin complex / oxygen transport / Scavenging of heme from plasma / endocytic vesicle lumen ...nitric oxide transport / hemoglobin alpha binding / cellular oxidant detoxification / hemoglobin binding / haptoglobin-hemoglobin complex / renal absorption / hemoglobin complex / oxygen transport / Scavenging of heme from plasma / endocytic vesicle lumen / blood vessel diameter maintenance / oxygen carrier activity / hydrogen peroxide catabolic process / carbon dioxide transport / response to hydrogen peroxide / Heme signaling / Erythrocytes take up oxygen and release carbon dioxide / Erythrocytes take up carbon dioxide and release oxygen / Cytoprotection by HMOX1 / Late endosomal microautophagy / oxygen binding / platelet aggregation / regulation of blood pressure / Chaperone Mediated Autophagy / positive regulation of nitric oxide biosynthetic process / tertiary granule lumen / Factors involved in megakaryocyte development and platelet production / blood microparticle / ficolin-1-rich granule lumen / iron ion binding / inflammatory response / heme binding / Neutrophil degranulation / extracellular space / extracellular exosome / extracellular region / metal ion binding / membrane / cytosol Similarity search - Function | ||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  MOLECULAR REPLACEMENT / Resolution: 2.25 Å MOLECULAR REPLACEMENT / Resolution: 2.25 Å | ||||||
 Authors Authors | Safo, M.K. / Abdulmalik, O. / Danso-Danquah, R. / Nokuri, S. / Joshi, G.S. / Musayev, F.N. / Asakura, T. / Abraham, D.J. | ||||||
 Citation Citation |  Journal: J.Med.Chem. / Year: 2004 Journal: J.Med.Chem. / Year: 2004Title: Structural basis for the potent antisickling effect of a novel class of five-membered heterocyclic aldehydic compounds Authors: Safo, M.K. / Abdulmalik, O. / Danso-Danquah, R. / Burnett, J.C. / Nokuri, S. / Joshi, G.S. / Musayev, F.N. / Asakura, T. / Abraham, D.J. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  1qxd.cif.gz 1qxd.cif.gz | 130.9 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb1qxd.ent.gz pdb1qxd.ent.gz | 104.3 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  1qxd.json.gz 1qxd.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/qx/1qxd https://data.pdbj.org/pub/pdb/validation_reports/qx/1qxd ftp://data.pdbj.org/pub/pdb/validation_reports/qx/1qxd ftp://data.pdbj.org/pub/pdb/validation_reports/qx/1qxd | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  1qxeC  1bbbS C: citing same article ( S: Starting model for refinement |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||
| 2 | 
| ||||||||||
| Unit cell |
| ||||||||||
| Components on special symmetry positions |
|
- Components
Components
-Protein , 2 types, 4 molecules ACBD
| #1: Protein | Mass: 15150.353 Da / Num. of mol.: 2 / Fragment: alpha chain / Source method: isolated from a natural source / Source: (natural)  Homo sapiens (human) / Cell: erythrocytes / Organ: blood / References: UniProt: P69905 Homo sapiens (human) / Cell: erythrocytes / Organ: blood / References: UniProt: P69905#2: Protein | Mass: 15890.198 Da / Num. of mol.: 2 / Fragment: beta chain / Source method: isolated from a natural source / Source: (natural)  Homo sapiens (human) / Cell: erythrocytes / Organ: blood / References: UniProt: P68871 Homo sapiens (human) / Cell: erythrocytes / Organ: blood / References: UniProt: P68871 |
|---|
-Non-polymers , 4 types, 314 molecules 

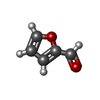




| #3: Chemical | ChemComp-SO4 / #4: Chemical | ChemComp-HEM / #5: Chemical | #6: Water | ChemComp-HOH / | |
|---|
-Details
| Has protein modification | Y |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.5 Å3/Da / Density % sol: 50.3 % | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Crystal grow | Temperature: 298 K / Method: liquid diffusion / pH: 6.5 Details: ammonium phosphate and sulfate, pH 6.5, LIQUID DIFFUSION, temperature 298K | ||||||||||||||||||
| Crystal grow | *PLUS pH: 6.4 / Method: other | ||||||||||||||||||
| Components of the solutions | *PLUS
|
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  ROTATING ANODE / Type: RIGAKU RU200 / Wavelength: 1.5418 Å ROTATING ANODE / Type: RIGAKU RU200 / Wavelength: 1.5418 Å |
| Detector | Type: RIGAKU RAXIS II / Detector: IMAGE PLATE / Date: May 13, 2002 / Details: mirrors |
| Radiation | Monochromator: osmic mirror / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1.5418 Å / Relative weight: 1 |
| Reflection | Resolution: 2.25→70 Å / Num. all: 32647 / Num. obs: 32647 / % possible obs: 97 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / Redundancy: 3.8 % / Biso Wilson estimate: 14.7 Å2 / Rmerge(I) obs: 0.075 / Net I/σ(I): 7 |
| Reflection shell | Resolution: 2.25→2.3 Å / Redundancy: 5 % / Rmerge(I) obs: 0.249 / Mean I/σ(I) obs: 1.8 / Num. unique all: 1991 / % possible all: 94.1 |
| Reflection | *PLUS Lowest resolution: 69.14 Å / % possible obs: 97 % / Num. measured all: 124853 |
| Reflection shell | *PLUS % possible obs: 94.1 % / Num. unique obs: 1991 / Num. measured obs: 9905 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: PDB entry 1BBB Resolution: 2.25→69.14 Å / Rfactor Rfree error: 0.007 / Isotropic thermal model: RESTRAINED / Cross valid method: THROUGHOUT / σ(F): 0 / σ(I): 0 / Stereochemistry target values: Engh & Huber
| |||||||||||||||||||||||||
| Solvent computation | Solvent model: FLAT MODEL / Bsol: 56.708 Å2 / ksol: 0.384124 e/Å3 | |||||||||||||||||||||||||
| Displacement parameters | Biso mean: 35.9 Å2
| |||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error free: 0.38 Å / Luzzati sigma a free: 0.6 Å | |||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 2.25→69.14 Å
| |||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||
| LS refinement shell | Resolution: 2.25→2.39 Å / Rfactor Rfree error: 0.024 / Total num. of bins used: 6
| |||||||||||||||||||||||||
| Xplor file |
| |||||||||||||||||||||||||
| Refinement | *PLUS % reflection Rfree: 5 % | |||||||||||||||||||||||||
| Solvent computation | *PLUS | |||||||||||||||||||||||||
| Displacement parameters | *PLUS | |||||||||||||||||||||||||
| Refine LS restraints | *PLUS
| |||||||||||||||||||||||||
| LS refinement shell | *PLUS Lowest resolution: 2.37 Å |
 Movie
Movie Controller
Controller












 PDBj
PDBj















