+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1prc | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CRYSTALLOGRAPHIC REFINEMENT AT 2.3 ANGSTROMS RESOLUTION AND REFINED MODEL OF THE PHOTOSYNTHETIC REACTION CENTER FROM RHODOPSEUDOMONAS VIRIDIS | |||||||||
 要素 要素 | (PHOTOSYNTHETIC REACTION ...) x 4 | |||||||||
 キーワード キーワード | PHOTOSYNTHETIC REACTION CENTER | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報plasma membrane-derived chromatophore membrane / plasma membrane light-harvesting complex / bacteriochlorophyll binding / photosynthetic electron transport in photosystem II / : / photosynthesis, light reaction / photosynthesis / electron transfer activity / iron ion binding / heme binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Blastochloris viridis (バクテリア) Blastochloris viridis (バクテリア) | |||||||||
| 手法 |  X線回折 / 解像度: 2.3 Å X線回折 / 解像度: 2.3 Å | |||||||||
 データ登録者 データ登録者 | Deisenhofer, J. / Epp, O. / Miki, K. / Huber, R. / Michel, H. | |||||||||
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 1995 ジャーナル: J.Mol.Biol. / 年: 1995タイトル: Crystallographic refinement at 2.3 A resolution and refined model of the photosynthetic reaction centre from Rhodopseudomonas viridis. 著者: Deisenhofer, J. / Epp, O. / Sinning, I. / Michel, H. #1:  ジャーナル: Science / 年: 1989 ジャーナル: Science / 年: 1989タイトル: The Photosynthetic Reaction Center from the Purple Bacterium Rhodopseudomonas Viridis 著者: Deisenhofer, J. / Michel, H. #2:  ジャーナル: Nature / 年: 1985 ジャーナル: Nature / 年: 1985タイトル: Structure of the Protein Subunits in the Photosynthetic Reaction Centre of Rhodopseudomonas Viridis at 3 Angstroms Resolution 著者: Deisenhofer, J. / Epp, O. / Miki, K. / Huber, R. / Michel, H. #3:  ジャーナル: J.Mol.Biol. / 年: 1984 ジャーナル: J.Mol.Biol. / 年: 1984タイトル: X-Ray Structure Analysis of a Membrane Protein Complex. Electron Density Map at 3 Angstroms Resolution and a Model of the Chromophores of the Photosynthetic Reaction Center from Rhodopseudomonas Viridis 著者: Deisenhofer, J. / Epp, O. / Miki, K. / Huber, R. / Michel, H. #4:  ジャーナル: J.Mol.Biol. / 年: 1982 ジャーナル: J.Mol.Biol. / 年: 1982タイトル: Three-Dimensional Crystals of a Membrane Protein Complex. The Photosynthetic Reaction Centre from Rhodopseudomonas Viridis 著者: Michel, H. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1prc.cif.gz 1prc.cif.gz | 280.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1prc.ent.gz pdb1prc.ent.gz | 218.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1prc.json.gz 1prc.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1prc_validation.pdf.gz 1prc_validation.pdf.gz | 3.3 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1prc_full_validation.pdf.gz 1prc_full_validation.pdf.gz | 3.4 MB | 表示 | |
| XML形式データ |  1prc_validation.xml.gz 1prc_validation.xml.gz | 64 KB | 表示 | |
| CIF形式データ |  1prc_validation.cif.gz 1prc_validation.cif.gz | 81 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/pr/1prc https://data.pdbj.org/pub/pdb/validation_reports/pr/1prc ftp://data.pdbj.org/pub/pdb/validation_reports/pr/1prc ftp://data.pdbj.org/pub/pdb/validation_reports/pr/1prc | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
| ||||||||
| Atom site foot note | 1: RESIDUES PRO C 6, PRO C 153, PRO C 330, PRO M 48, PRO H 42, AND PRO H 79 ARE CIS PROLINES. 2: SEE REMARK 4. | ||||||||
| Components on special symmetry positions |
|
- 要素
要素
-PHOTOSYNTHETIC REACTION ... , 4種, 4分子 CLMH
| #1: タンパク質 | 分子量: 37450.801 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Blastochloris viridis (バクテリア) Blastochloris viridis (バクテリア)参照: UniProt: P07173 |
|---|---|
| #2: タンパク質 | 分子量: 30469.104 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Blastochloris viridis (バクテリア) Blastochloris viridis (バクテリア)参照: UniProt: P06009 |
| #3: タンパク質 | 分子量: 35932.188 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Blastochloris viridis (バクテリア) Blastochloris viridis (バクテリア)参照: UniProt: P06010 |
| #4: タンパク質 | 分子量: 28557.453 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Blastochloris viridis (バクテリア) Blastochloris viridis (バクテリア)参照: UniProt: P06008 |
-非ポリマー , 10種, 224分子 
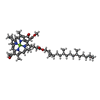
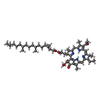
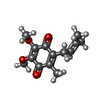















| #5: 化合物 | ChemComp-HEC / #6: 化合物 | ChemComp-BCB / #7: 化合物 | #8: 化合物 | ChemComp-UQ1 / | #9: 化合物 | ChemComp-FE / | #10: 化合物 | ChemComp-SO4 / #11: 化合物 | ChemComp-MQ7 / | #12: 化合物 | ChemComp-NS1 / | #13: 化合物 | #14: 水 | ChemComp-HOH / | |
|---|
-詳細
| Has protein modification | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 5.36 Å3/Da / 溶媒含有率: 77.04 % | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | *PLUS pH: 6 / 手法: 蒸気拡散法 / 詳細: Michel, H., (1982) J.Mol.Biol., 158, 567. | |||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 放射 | 散乱光タイプ: x-ray |
|---|---|
| 放射波長 | 相対比: 1 |
| 反射 | *PLUS 最高解像度: 2.3 Å / Num. obs: 95762 / Observed criterion σ(I): 2 / Rmerge(I) obs: 0.091 |
| 反射 シェル | *PLUS 最高解像度: 2.3 Å / 最低解像度: 2.4 Å / % possible obs: 49.2 % |
- 解析
解析
| ソフトウェア | 名称: TNT / 分類: 精密化 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 2.3→19.99 Å /
| ||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.26 Å | ||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.3→19.99 Å
| ||||||||||||
| 精密化 | *PLUS Rfactor obs: 0.193 | ||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||
| 原子変位パラメータ | *PLUS Biso mean: 22.5 Å2 |
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj




















