+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1ppk | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CRYSTALLOGRAPHIC ANALYSIS OF TRANSITION STATE MIMICS BOUND TO PENICILLOPEPSIN: PHOSPHOROUS-CONTAINING PEPTIDE ANALOGUES | |||||||||
 要素 要素 | PENICILLOPEPSIN | |||||||||
 キーワード キーワード | HYDROLASE/HYDROLASE INHIBITOR / ACID PROTEINASE / HYDROLASE-HYDROLASE INHIBITOR complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報penicillopepsin / aspartic-type endopeptidase activity / proteolysis / extracellular region 類似検索 - 分子機能 | |||||||||
| 生物種 |  Penicillium janthinellum (菌類) Penicillium janthinellum (菌類) | |||||||||
| 手法 |  X線回折 / 解像度: 1.8 Å X線回折 / 解像度: 1.8 Å | |||||||||
 データ登録者 データ登録者 | Strynadka, N.C.J. / James, M.N.G. | |||||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 1992 ジャーナル: Biochemistry / 年: 1992タイトル: Crystallographic analysis of transition-state mimics bound to penicillopepsin: phosphorus-containing peptide analogues. 著者: Fraser, M.E. / Strynadka, N.C. / Bartlett, P.A. / Hanson, J.E. / James, M.N. #1:  ジャーナル: Biochemistry / 年: 1992 ジャーナル: Biochemistry / 年: 1992タイトル: Crystallographic Analysis of Transition State Mimics Bound to Penicillopepsin: Difluorostatine-and Difluorostatone-Containing Peptides 著者: James, M.N.G. / Sielecki, A.R. / Hayakawa, K. / Gelb, M.H. #2:  ジャーナル: Biological Macromolecules and Assemblies / 年: 1987 ジャーナル: Biological Macromolecules and Assemblies / 年: 1987タイトル: Aspartic Proteinases and Their Catalytic Pathway 著者: James, M.N.G. / Sielecki, A.R. #3:  ジャーナル: Biochemistry / 年: 1985 ジャーナル: Biochemistry / 年: 1985タイトル: Stereochemical Analysis of Peptide Bond Hydrolysis Catalyzed by the Aspartic Proteinase Penicillopepsin 著者: James, M.N.G. / Sielecki, A.R. #4:  ジャーナル: Aspartic Proteinases and Their Inhibitors / 年: 1985 ジャーナル: Aspartic Proteinases and Their Inhibitors / 年: 1985タイトル: X-Ray Diffraction Studies on Penicillopepsin and its Complexes: The Hydrolytic Mechanism 著者: James, M.N.G. / Sielecki, A.R. / Hofmann, T. #5:  ジャーナル: Biochemistry / 年: 1984 ジャーナル: Biochemistry / 年: 1984タイトル: Effect of Ph on the Activities of Penicillopepsin and Rhizopus Pepsin and a Proposal for the Productive Substrate Binding Mode in Penicillopepsin 著者: Hofmann, T. / Hodges, R.S. / James, M.N.G. #6:  ジャーナル: Peptides: Structure and Function, Proceedings of the of the Eighth American Peptide Symposium ジャーナル: Peptides: Structure and Function, Proceedings of the of the Eighth American Peptide Symposium年: 1983 タイトル: Crystallographic Analysis of a Pepstatin Analogue Binding to the Aspartyl Proteinase Penicillopepsin at 1.8 Angstroms Resolution 著者: James, M.N.G. / Sielecki, A.R. / Moult, J. #7:  ジャーナル: J.Mol.Biol. / 年: 1983 ジャーナル: J.Mol.Biol. / 年: 1983タイトル: Structure and Refinement of Penicillopepsin at 1.8 Angstroms Resolution 著者: James, M.N.G. / Sielecki, A.R. #8:  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1982 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1982タイトル: Conformational Flexibility in the Active Sites of Aspartyl Proteinases Revealed by a Pepstatin Fragment Binding to Penicillopepsin 著者: James, M.N.G. / Sielecki, A. / Salituro, F. / Rich, D.H. / Hofmann, T. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1ppk.cif.gz 1ppk.cif.gz | 81.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1ppk.ent.gz pdb1ppk.ent.gz | 59.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1ppk.json.gz 1ppk.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1ppk_validation.pdf.gz 1ppk_validation.pdf.gz | 496.7 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1ppk_full_validation.pdf.gz 1ppk_full_validation.pdf.gz | 503.6 KB | 表示 | |
| XML形式データ |  1ppk_validation.xml.gz 1ppk_validation.xml.gz | 9.3 KB | 表示 | |
| CIF形式データ |  1ppk_validation.cif.gz 1ppk_validation.cif.gz | 15.2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/pp/1ppk https://data.pdbj.org/pub/pdb/validation_reports/pp/1ppk ftp://data.pdbj.org/pub/pdb/validation_reports/pp/1ppk ftp://data.pdbj.org/pub/pdb/validation_reports/pp/1ppk | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
| ||||||||
| Atom site foot note | 1: CIS PROLINE - PRO E 134 / 2: THE REGION FROM SER E 277 - SER E 281 IS POORLY ORDERED. / 3: CIS PROLINE - PRO E 315 | ||||||||
| Components on special symmetry positions |
|
- 要素
要素
-タンパク質 , 1種, 1分子 E
| #1: タンパク質 | 分子量: 33468.809 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Penicillium janthinellum (菌類) / 参照: UniProt: P00798, penicillopepsin Penicillium janthinellum (菌類) / 参照: UniProt: P00798, penicillopepsin |
|---|
-糖 , 2種, 2分子 
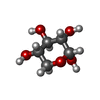

| #3: 糖 | ChemComp-MAN / |
|---|---|
| #4: 糖 | ChemComp-HSY / |
-非ポリマー , 4種, 278分子 

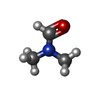


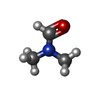

| #2: 化合物 | ChemComp-IVV / |
|---|---|
| #5: 化合物 | ChemComp-SO4 / |
| #6: 化合物 | ChemComp-DMF / |
| #7: 水 | ChemComp-HOH / |
-詳細
| Has protein modification | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 1.99 Å3/Da / 溶媒含有率: 38.2 % | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | *PLUS 温度: 18-22 ℃ / pH: 4.4 / 手法: 蒸気拡散法, ハンギングドロップ法 | ||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 放射 | 散乱光タイプ: x-ray |
|---|---|
| 放射波長 | 相対比: 1 |
| 反射 | 最高解像度: 1.8 Å |
| 反射 | *PLUS 最高解像度: 1.8 Å / 最低解像度: 45 Å / Num. obs: 24917 / Num. measured all: 29429 / Rmerge(I) obs: 0.033 |
- 解析
解析
| ソフトウェア | 名称: PROLSQ / 分類: 精密化 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 1.8→8 Å / σ(F): 3 /
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.8→8 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: PROLSQ / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS Rfactor obs: 0.132 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS |
 ムービー
ムービー コントローラー
コントローラー

























 PDBj
PDBj








