| 登録情報 | データベース: PDB / ID: 1p83
|
|---|
| タイトル | NMR STRUCTURE OF 1-25 FRAGMENT OF MYCOBACTERIUM TUBERCULOSIS CPN10 |
|---|
 要素 要素 | 10 kDa chaperonin |
|---|
 キーワード キーワード | CHAPERONE / Cpn10 / Mycobacterium tuberculosis / Helix formation |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
cell wall / zymogen binding / chaperone cofactor-dependent protein refolding / protein folding chaperone / peptidoglycan-based cell wall / unfolded protein binding / protein-folding chaperone binding / cellular response to heat / response to heat / response to antibiotic ...cell wall / zymogen binding / chaperone cofactor-dependent protein refolding / protein folding chaperone / peptidoglycan-based cell wall / unfolded protein binding / protein-folding chaperone binding / cellular response to heat / response to heat / response to antibiotic / regulation of DNA-templated transcription / ATP hydrolysis activity / extracellular region / ATP binding / metal ion binding / plasma membrane / cytosol類似検索 - 分子機能 Chaperonin GroES, conserved site / Chaperonins cpn10 signature. / Chaperonin 10 Kd subunit / GroES chaperonin family / GroES chaperonin superfamily / Chaperonin 10 Kd subunit / GroES-like superfamily類似検索 - ドメイン・相同性 |
|---|
| 手法 | 溶液NMR / molecular dynamics |
|---|
 データ登録者 データ登録者 | Ciutti, A. / Spiga, O. / Giannozzi, E. / Scarselli, M. / Di Maro, D. / Calamandrei, D. / Niccolai, N. / Bernini, A. |
|---|
 引用 引用 | #1:  ジャーナル: To be Publishedタイトル ジャーナル: To be Publishedタイトル: Mycobacterium tuberculosis chaperonin 10 is secreted in the macrophage phagolysosome: Is secretion due to dissociation and the adoption of partially helical structure at the membrane 著者: Fossati, G. / Izzo, G. / Rizzi, E. / Gancia, E. / Modena, D. / Moras, M.L. / Niccolai, N. / Giannozzi, E. / Spiga, O. / Bono, L. / Marone, P. / Leone, E. / Mangili, F. / Harding, S. / ...著者: Fossati, G. / Izzo, G. / Rizzi, E. / Gancia, E. / Modena, D. / Moras, M.L. / Niccolai, N. / Giannozzi, E. / Spiga, O. / Bono, L. / Marone, P. / Leone, E. / Mangili, F. / Harding, S. / Errington, N. / Walter, C. / Henderson, B. / Roberts, M.M. / Coates, A.R.M. / Casetta, B. / Mascagni, P. |
|---|
| 履歴 | | 登録 | 2003年5月6日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2003年5月27日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年4月29日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2022年2月23日 | Group: Data collection / Database references / Derived calculations
カテゴリ: database_2 / pdbx_nmr_software ...database_2 / pdbx_nmr_software / pdbx_struct_assembly / pdbx_struct_oper_list
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_software.name |
|---|
| 改定 1.4 | 2024年5月22日 | Group: Data collection / カテゴリ: chem_comp_atom / chem_comp_bond |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 データ登録者
データ登録者 引用
引用 ジャーナル: To be Published
ジャーナル: To be Published ジャーナル: To be Published
ジャーナル: To be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1p83.cif.gz
1p83.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1p83.ent.gz
pdb1p83.ent.gz PDB形式
PDB形式 1p83.json.gz
1p83.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 1p83_validation.pdf.gz
1p83_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 1p83_full_validation.pdf.gz
1p83_full_validation.pdf.gz 1p83_validation.xml.gz
1p83_validation.xml.gz 1p83_validation.cif.gz
1p83_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/p8/1p83
https://data.pdbj.org/pub/pdb/validation_reports/p8/1p83 ftp://data.pdbj.org/pub/pdb/validation_reports/p8/1p83
ftp://data.pdbj.org/pub/pdb/validation_reports/p8/1p83 リンク
リンク 集合体
集合体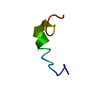
 要素
要素 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj

 Amber
Amber