ソフトウェア 名称 バージョン 分類 5.1.24精密化 データ削減 データスケーリング 位相決定
精密化 構造決定の手法 開始モデル 解像度 / Cor.coef. Fo :Fc / Cor.coef. Fo :Fc free / SU B / SU ML / TLS residual ADP flag Isotropic thermal model 交差検証法 / σ(F) / ESU R / ESU R Free / 立体化学のターゲット値 Rfactor 反射数 %反射 Selection details Rfree 0.27589 677 4.6 % RANDOM Rwork 0.23908 - - - obs 0.24089 13935 85.16 % - all - 17158 - -
溶媒の処理 イオンプローブ半径 / 減衰半径 / VDWプローブ半径 / 溶媒モデル 原子変位パラメータ Biso mean 2 Baniso -1 Baniso -2 Baniso -3 1- -0.6 Å2 0 Å2 0 Å2 2- - 2.99 Å2 0 Å2 3- - - -2.39 Å2
精密化ステップ サイクル / 解像度 タンパク質 核酸 リガンド 溶媒 全体 原子数 1384 812 0 94 2290
拘束条件 大きな表を表示 (5 x 25) 大きな表を隠す Refine-ID タイプ Dev ideal Dev ideal target 数 X-RAY DIFFRACTION r_bond_refined_d0.013 0.021 2301 X-RAY DIFFRACTION r_bond_other_dX-RAY DIFFRACTION r_angle_refined_deg1.625 2.415 3269 X-RAY DIFFRACTION r_angle_other_degX-RAY DIFFRACTION r_dihedral_angle_1_deg4.748 5 182 X-RAY DIFFRACTION r_dihedral_angle_2_degX-RAY DIFFRACTION r_chiral_restr0.081 0.2 337 X-RAY DIFFRACTION r_gen_planes_refined0.006 0.02 1434 X-RAY DIFFRACTION r_gen_planes_otherX-RAY DIFFRACTION r_nbd_refined0.227 0.2 950 X-RAY DIFFRACTION r_nbd_otherX-RAY DIFFRACTION r_nbtor_otherX-RAY DIFFRACTION r_xyhbond_nbd_refined0.168 0.2 137 X-RAY DIFFRACTION r_xyhbond_nbd_otherX-RAY DIFFRACTION r_symmetry_vdw_refined0.253 0.2 52 X-RAY DIFFRACTION r_symmetry_vdw_otherX-RAY DIFFRACTION r_symmetry_hbond_refined0.024 0.2 2 X-RAY DIFFRACTION r_symmetry_hbond_otherX-RAY DIFFRACTION r_mcbond_it0.421 1.5 915 X-RAY DIFFRACTION r_mcangle_it0.787 2 1467 X-RAY DIFFRACTION r_scbond_it1.688 3 1386 X-RAY DIFFRACTION r_scangle_it2.55 4.5 1802 X-RAY DIFFRACTION r_rigid_bond_restrX-RAY DIFFRACTION r_sphericity_freeX-RAY DIFFRACTION r_sphericity_bonded
Refine LS restraints NCS Dom-ID / Auth asym-ID / Ens-ID / 数 / Refine-ID
タイプ Rms dev position (Å)Weight position medium positional0.14 0.5 medium thermal0.38 2
LS精密化 シェル 解像度 / Total num. of bins used / Rfactor 反射数 Rfree 0.411 47 Rwork 0.336 1235
精密化 TLS 手法 / Refine-ID
大きな表を表示 (25 x 6) 大きな表を隠す ID L11 (°2 )L12 (°2 )L13 (°2 )L22 (°2 )L23 (°2 )L33 (°2 )S11 (Å °)S12 (Å °)S13 (Å °)S21 (Å °)S22 (Å °)S23 (Å °)S31 (Å °)S32 (Å °)S33 (Å °)T11 (Å2 )T12 (Å2 )T13 (Å2 )T22 (Å2 )T23 (Å2 )T33 (Å2 )Origin x (Å)Origin y (Å)Origin z (Å)1 4.4402 0.1257 0.0837 3.2119 0.204 4.3348 -0.1221 -1.121 -0.1711 0.5365 0.0179 -0.0808 0.1669 0.1138 0.1042 0.1868 0.0803 0.0128 0.5175 0.08 0.2113 19.214 15.2371 88.7704 2 2.3716 -2.0081 -0.665 7.1912 -3.777 6.6672 0.1143 -0.0642 0.089 -0.3651 -0.2105 -0.1928 -0.1782 0.3405 0.0963 0.0604 -0.0311 -0.0647 0.1519 -0.013 0.1151 32.048 28.3045 64.8618 3 2.5142 -2.5451 -0.6479 4.2805 3.9834 10.8624 -0.1048 -0.0151 -0.0653 -0.2007 0.1531 0.0621 -0.0778 -0.0547 -0.0483 0.1032 -0.0119 0.0138 0.1447 0.0315 0.102 6.6169 13.3915 62.2819 4 4.5763 -1.4454 -2.6599 8.7232 0.2857 8.6714 -0.0145 -0.3976 0.5962 0.8406 -0.0062 0.4825 0.2479 -0.8986 0.0206 0.281 -0.036 0.0419 0.2367 -0.1801 0.2414 22.03 41.0978 70.8414 5 4.3519 1.9439 0.5927 5.6616 -0.3558 -0.9205 -0.098 -0.1792 -0.3681 -0.1545 -0.2495 -0.8097 0.1948 -0.0655 0.3475 0.2579 -0.045 0.0009 0.0711 -0.0525 0.1312 20.6171 21.2507 60.8879 6 7.2716 -4.0158 6.9926 9.7416 -3.963 20.7696 0.266 -0.4629 -1.1317 -0.1767 -0.1019 -0.0239 1.4658 0.9843 -0.164 0.6124 -0.0246 -0.0126 0.2616 0.1875 0.3302 14.5611 -1.3294 64.0045
精密化 TLSグループ ID Refine-ID Refine TLS-ID Auth asym-ID Auth seq-ID 1 X-RAY DIFFRACTION 1 A1 - 52 2 X-RAY DIFFRACTION 1 B1 - 52 3 X-RAY DIFFRACTION 1 A77 - 92 4 X-RAY DIFFRACTION 1 B77 - 92 5 X-RAY DIFFRACTION 2 A53 - 76 6 X-RAY DIFFRACTION 3 B53 - 76 7 X-RAY DIFFRACTION 4 C1 - 5 8 X-RAY DIFFRACTION 4 D16 - 20 9 X-RAY DIFFRACTION 5 C6 - 15 10 X-RAY DIFFRACTION 5 D5 - 15 11 X-RAY DIFFRACTION 6 C16 - 20 12 X-RAY DIFFRACTION 6 D1 - 4
精密化 *PLUS
最高解像度 / 最低解像度 / % reflection Rfree / Rfactor Rfree / Rfactor Rwork 溶媒の処理 *PLUS
原子変位パラメータ *PLUS
拘束条件 *PLUS
Refine-ID タイプ Dev ideal X-RAY DIFFRACTION r_bond_d0.013 X-RAY DIFFRACTION r_angle_dX-RAY DIFFRACTION r_angle_deg1.625
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Anabaena sp. (バクテリア)
Anabaena sp. (バクテリア) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  フーリエ合成 / 解像度: 2.25 Å
フーリエ合成 / 解像度: 2.25 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Embo J. / 年: 2003
ジャーナル: Embo J. / 年: 2003 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1p78.cif.gz
1p78.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1p78.ent.gz
pdb1p78.ent.gz PDB形式
PDB形式 1p78.json.gz
1p78.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 1p78_validation.pdf.gz
1p78_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 1p78_full_validation.pdf.gz
1p78_full_validation.pdf.gz 1p78_validation.xml.gz
1p78_validation.xml.gz 1p78_validation.cif.gz
1p78_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/p7/1p78
https://data.pdbj.org/pub/pdb/validation_reports/p7/1p78 ftp://data.pdbj.org/pub/pdb/validation_reports/p7/1p78
ftp://data.pdbj.org/pub/pdb/validation_reports/p7/1p78 リンク
リンク 集合体
集合体
 要素
要素 Anabaena sp. (バクテリア) / 遺伝子: HUP OR HANA OR ASR3935 / プラスミド: pET21A (pETAHU) / 発現宿主:
Anabaena sp. (バクテリア) / 遺伝子: HUP OR HANA OR ASR3935 / プラスミド: pET21A (pETAHU) / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 19-ID / 波長: 1.0078 Å
/ ビームライン: 19-ID / 波長: 1.0078 Å 解析
解析 フーリエ合成
フーリエ合成 ムービー
ムービー コントローラー
コントローラー




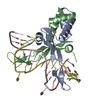



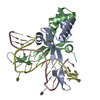
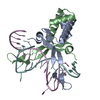







 PDBj
PDBj







































