+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1p02 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | STRUCTURE ANALYSIS OF SPECIFICITY. ALPHA-LYTIC PROTEASE COMPLEXES WITH ANALOGUES OF REACTION INTERMEDIATES | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | HYDROLASE/HYDROLASE INHIBITOR / HYDROLASE-HYDROLASE INHIBITOR COMPLEX | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報alpha-lytic endopeptidase / serine-type endopeptidase activity / proteolysis / extracellular region 類似検索 - 分子機能 | ||||||
| 生物種 |  Lysobacter enzymogenes (バクテリア) Lysobacter enzymogenes (バクテリア) | ||||||
| 手法 |  X線回折 / 解像度: 2 Å X線回折 / 解像度: 2 Å | ||||||
 データ登録者 データ登録者 | Bone, R. / Agard, D.A. | ||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 1989 ジャーナル: Biochemistry / 年: 1989タイトル: Structural analysis of specificity: alpha-lytic protease complexes with analogues of reaction intermediates. 著者: Bone, R. / Frank, D. / Kettner, C.A. / Agard, D.A. #1:  ジャーナル: To be Published ジャーナル: To be Publishedタイトル: Structural Plasticity as a Determinant of Enzyme Specificity. Creating Broadly Specific Proteases 著者: Bone, R. / Silen, J.L. / Agard, D.A. #2:  ジャーナル: Biochemistry / 年: 1988 ジャーナル: Biochemistry / 年: 1988タイトル: Kinetic Properties of the Binding of Alpha-Lytic Protease to Peptide Boronic Acids 著者: Kettner, C.A. / Bone, R. / Agard, D.A. / Bachovchin, W.W. #3:  ジャーナル: Biochemistry / 年: 1987 ジャーナル: Biochemistry / 年: 1987タイトル: Serine Protease Mechanism. Structure of an Inhibitory Complex of Alpha-Lytic Protease and a Tightly Bound Peptide Boronic Acid 著者: Bone, R. / Shenvi, A.B. / Kettner, C.A. / Agard, D.A. #4:  ジャーナル: J.Mol.Biol. / 年: 1985 ジャーナル: J.Mol.Biol. / 年: 1985タイトル: Refined Structure of Alpha-Lytic Protease at 1.7 Angstroms Resolution. Analysis of Hydrogen Bonding and Solvent Structure 著者: Fujinaga, M. / Delbaere, L.T.J. / Brayer, G.D. / James, M.N.G. #5:  ジャーナル: J.Mol.Biol. / 年: 1979 ジャーナル: J.Mol.Biol. / 年: 1979タイトル: Molecular Structure of the Alpha-Lytic Protease from Myxobacter 495 at 2.8 Angstroms Resolution 著者: Brayer, G.D. / Delbaere, L.T.J. / James, M.N.G. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1p02.cif.gz 1p02.cif.gz | 53.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1p02.ent.gz pdb1p02.ent.gz | 37.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1p02.json.gz 1p02.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1p02_validation.pdf.gz 1p02_validation.pdf.gz | 384.9 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1p02_full_validation.pdf.gz 1p02_full_validation.pdf.gz | 393.1 KB | 表示 | |
| XML形式データ |  1p02_validation.xml.gz 1p02_validation.xml.gz | 6.5 KB | 表示 | |
| CIF形式データ |  1p02_validation.cif.gz 1p02_validation.cif.gz | 9.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/p0/1p02 https://data.pdbj.org/pub/pdb/validation_reports/p0/1p02 ftp://data.pdbj.org/pub/pdb/validation_reports/p0/1p02 ftp://data.pdbj.org/pub/pdb/validation_reports/p0/1p02 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 19875.131 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Lysobacter enzymogenes (バクテリア) Lysobacter enzymogenes (バクテリア)参照: UniProt: P00778, alpha-lytic endopeptidase |
|---|---|
| #2: タンパク質・ペプチド | |
| #3: 化合物 | ChemComp-SO4 / |
| #4: 水 | ChemComp-HOH / |
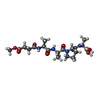
| 構成要素の詳細 | INHIBITORY PEPTIDE BORONIC ACIDS ARE PEPTIDE ANALOGS IN WHICH THE C-TERMINAL CARBOXYL GROUP HAS ...INHIBITORY |
| Has protein modification | Y |
| 非ポリマーの詳細 | INHIBITORY PEPTIDE BORONIC ACIDS ARE PEPTIDE ANALOGUES IN WHICH THE C-TERMINAL CARBOXY GROUP (COOH) ...INHIBITORY |
| 配列の詳細 | CHAIN A RESIDUE NUMBERING IS DONE BY HOMOLOGY WITH CHYMOTRYPSIN FOR RESIDUES 15A - 244 AS DESCRIBED ...CHAIN A RESIDUE NUMBERING IS DONE BY HOMOLOGY WITH CHYMOTRYPS |
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.52 Å3/Da / 溶媒含有率: 51.28 % | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | *PLUS pH: 6.8 / 手法: 蒸気拡散法, ハンギングドロップ法詳細: seeding, refer to Bone, R. et al (1987). Biochemistry, 27, 7609-7614. | ||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 放射 | 散乱光タイプ: x-ray |
|---|---|
| 放射波長 | 相対比: 1 |
| 反射 | 最高解像度: 2 Å |
| 反射 | *PLUS % possible obs: 88 % |
- 解析
解析
| ソフトウェア | 名称: PROLSQ / 分類: 精密化 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | Rfactor obs: 0.147 / 最高解像度: 2 Å 詳細: THE METHOXYSUCCINYL PORTION OF THE INHIBITOR WAS DISORDERED AND NO COORDINATES ARE INCLUDED FOR IT IN THIS ENTRY | ||||||||||||
| 精密化ステップ | サイクル: LAST / 最高解像度: 2 Å
| ||||||||||||
| 精密化 | *PLUS σ(I): 3 / 最高解像度: 2 Å / Rfactor obs: 0.147 | ||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||
| 原子変位パラメータ | *PLUS | ||||||||||||
| 拘束条件 | *PLUS タイプ: p_bond_d / Dev ideal: 0.12 |
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj