+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1mwj | ||||||
|---|---|---|---|---|---|---|---|
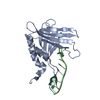
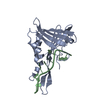
| タイトル | Crystal Structure of a MUG-DNA pseudo substrate complex | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | HYDROLASE/DNA / Rossmann Fold / non-hydrolysable DNA-complex / Uracil recognition. / HYDROLASE-DNA COMPLEX | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報double-stranded uracil-DNA glycosylase / pyrimidine-specific mismatch base pair DNA N-glycosylase activity / base-excision repair, AP site formation / uracil DNA N-glycosylase activity / DNA binding / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.85 Å 分子置換 / 解像度: 2.85 Å | ||||||
 データ登録者 データ登録者 | Barrett, T.E. / Scharer, O. / Savva, R. / Brown, T. / Jiricny, J. / Verdine, G.L. / Pearl, L.H. | ||||||
 引用 引用 |  ジャーナル: Embo J. / 年: 1999 ジャーナル: Embo J. / 年: 1999タイトル: Crystal Structure of a thwarted mismatch glycosylase DNA repair complex 著者: Barrett, T.E. / Scharer, O. / Savva, R. / Brown, T. / Jiricny, J. / Verdine, G.L. / Pearl, L.H. | ||||||
| 履歴 |
| ||||||
| Remark 300 | BIOMOLECULE THIS ENTRY CONTAINS THE CRYSTALLOGRAPHIC ASYMMETRIC UNIT WHICH CONSISTS OF 2 CHAIN(S). ...BIOMOLECULE THIS ENTRY CONTAINS THE CRYSTALLOGRAPHIC ASYMMETRIC UNIT WHICH CONSISTS OF 2 CHAIN(S). The second strand of the DNA molecule is generated by applying symmetry operator 7_555 : y, x, -z, to DU and bases 8-12, and applying symmetry operator 7_554 : y, x, -1-z, to bases 1-6. |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1mwj.cif.gz 1mwj.cif.gz | 54.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1mwj.ent.gz pdb1mwj.ent.gz | 36.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1mwj.json.gz 1mwj.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1mwj_validation.pdf.gz 1mwj_validation.pdf.gz | 370.1 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1mwj_full_validation.pdf.gz 1mwj_full_validation.pdf.gz | 372.7 KB | 表示 | |
| XML形式データ |  1mwj_validation.xml.gz 1mwj_validation.xml.gz | 5.2 KB | 表示 | |
| CIF形式データ |  1mwj_validation.cif.gz 1mwj_validation.cif.gz | 7.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/mw/1mwj https://data.pdbj.org/pub/pdb/validation_reports/mw/1mwj ftp://data.pdbj.org/pub/pdb/validation_reports/mw/1mwj ftp://data.pdbj.org/pub/pdb/validation_reports/mw/1mwj | HTTPS FTP |
-関連構造データ
| 関連構造データ | |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: DNA鎖 | 分子量: 3665.365 Da / 分子数: 1 / 由来タイプ: 合成 |
|---|---|
| #2: タンパク質 | 分子量: 18696.352 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   参照: UniProt: P0A9H1, 加水分解酵素; 糖加水分解酵素; N-グリコシル化合物加水分解酵素 |
| #3: 水 | ChemComp-HOH / |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.6 Å3/Da / 溶媒含有率: 52.78 % | |||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 温度: 289 K / 手法: マイクロバッチ法 / pH: 4.6 詳細: PEG4000, Sodium Acetate, pH 4.6, Microbatch, temperature 289K | |||||||||||||||||||||||||||||||||||
| 溶液の組成 |
| |||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 手法: microdialysis / 詳細: Barrett, T.E., (1998) Cell., 92, 117. | |||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SRS SRS  / ビームライン: PX7.2 / 波長: 1.488 Å / ビームライン: PX7.2 / 波長: 1.488 Å |
| 検出器 | タイプ: MARRESEARCH / 検出器: IMAGE PLATE / 日付: 1998年2月15日 |
| 放射 | モノクロメーター: Si 111 CHANNEL / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.488 Å / 相対比: 1 |
| 反射 | 解像度: 2.85→20 Å / Num. all: 5478 / Num. obs: 5478 / % possible obs: 94.5 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 2 / 冗長度: 2.3 % / Biso Wilson estimate: 38.2 Å2 / Rmerge(I) obs: 0.087 / Rsym value: 0.087 / Net I/σ(I): 9.7 |
| 反射 シェル | 解像度: 2.85→2.95 Å / 冗長度: 1.8 % / Rmerge(I) obs: 0.263 / Mean I/σ(I) obs: 2 / Num. unique all: 472 / Rsym value: 0.263 / % possible all: 85.7 |
| 反射 | *PLUS Num. obs: 5479 / Rmerge(I) obs: 0.087 |
| 反射 シェル | *PLUS % possible obs: 96.7 % / Rmerge(I) obs: 0.278 / Mean I/σ(I) obs: 2.9 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: Native MuG 解像度: 2.85→14.97 Å / Rfactor Rfree error: 0.017 / Data cutoff high absF: 1104323.12 / Data cutoff low absF: 0 / Isotropic thermal model: RESTRAINED / 交差検証法: THROUGHOUT / σ(F): 0 / 立体化学のターゲット値: Engh & Huber 詳細: NO ELECTRON DENSITY IS OBSERVED FOR RESDUES 166 TO 168 RESIDUES A1, A109, A150 AND A151 WERE REFINED AS ALANINE
| ||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 22.3 Å2 | ||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.35 Å / Luzzati d res low obs: 5 Å / Luzzati sigma a obs: 0.38 Å | ||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.85→14.97 Å
| ||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.85→2.93 Å / Rfactor Rfree error: 0.018 / Total num. of bins used: 8
| ||||||||||||||||||||||||||||||||||||
| Xplor file |
| ||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称:  XPLOR / バージョン: 3.851 / 分類: refinement XPLOR / バージョン: 3.851 / 分類: refinement | ||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS 最低解像度: 20 Å / Rfactor obs: 0.23 / Rfactor Rfree: 0.25 / Rfactor Rwork: 0.19 | ||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | ||||||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
| ||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | *PLUS Rfactor Rfree: 0.367 / Rfactor Rwork: 0.295 |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj







































