[English] 日本語
 Yorodumi
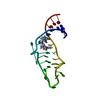
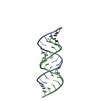
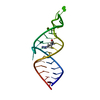
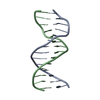
Yorodumi- PDB-1ldz: SOLUTION STRUCTURE OF THE LEAD-DEPENDENT RIBOZYME, NMR, 25 STRUCTURES -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 1ldz | ||||||
|---|---|---|---|---|---|---|---|
| Title | SOLUTION STRUCTURE OF THE LEAD-DEPENDENT RIBOZYME, NMR, 25 STRUCTURES | ||||||
 Components Components | LEAD-DEPENDENT RIBOZYME | ||||||
 Keywords Keywords | RNA / CATALYTIC RNA / INTERNAL LOOPS / LEADZYME / NMR SPECTROSCOPY / RNA STRUCTURE | ||||||
| Function / homology | RNA / RNA (> 10) Function and homology information Function and homology information | ||||||
| Method | SOLUTION NMR / SIMULATED ANNEALING FROM RANDOM TORSION ANGLES | ||||||
 Authors Authors | Hoogstraten, C.G. / Legault, P. / Pardi, A. | ||||||
 Citation Citation |  Journal: J.Mol.Biol. / Year: 1998 Journal: J.Mol.Biol. / Year: 1998Title: NMR solution structure of the lead-dependent ribozyme: evidence for dynamics in RNA catalysis. Authors: Hoogstraten, C.G. / Legault, P. / Pardi, A. #1:  Journal: J.Mol.Biol. / Year: 1998 Journal: J.Mol.Biol. / Year: 1998Title: Order, Dynamics and Metal-Binding in the Lead-Dependent Ribozyme Authors: Legault, P. / Hoogstraten, C.G. / Metlitzky, E. / Pardi, A. #2:  Journal: Biochemistry / Year: 1994 Journal: Biochemistry / Year: 1994Title: Properties of an in Vitro Selected Pb2+ Cleavage Motif Authors: Pan, T. / Dichtl, B. / Uhlenbeck, O.C. #3:  Journal: Nature / Year: 1992 Journal: Nature / Year: 1992Title: A Small Metalloribozyme with a Two-Step Mechanism Authors: Pan, T. / Uhlenbeck, O.C. #4:  Journal: Biochemistry / Year: 1992 Journal: Biochemistry / Year: 1992Title: In Vitro Selection of Rnas that Undergo Autolytic Cleavage with Pb2+ Authors: Pan, T. / Uhlenbeck, O.C. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  1ldz.cif.gz 1ldz.cif.gz | 465.8 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb1ldz.ent.gz pdb1ldz.ent.gz | 394.6 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  1ldz.json.gz 1ldz.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/ld/1ldz https://data.pdbj.org/pub/pdb/validation_reports/ld/1ldz ftp://data.pdbj.org/pub/pdb/validation_reports/ld/1ldz ftp://data.pdbj.org/pub/pdb/validation_reports/ld/1ldz | HTTPS FTP |
|---|
-Related structure data
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR ensembles |
|
- Components
Components
| #1: RNA chain | Mass: 9761.906 Da / Num. of mol.: 1 / Source method: obtained synthetically Details: PREPARED BY IN VITRO TRANSCRIPTION FROM SYNTHETIC DNA TEMPLATE |
|---|
-Experimental details
-Experiment
| Experiment | Method: SOLUTION NMR | ||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR experiment |
| ||||||||||||||||||||||||||||||||||||||||||||
| NMR details | Text: THE STRUCTURE WAS DETERMINED FROM DISTANCE AND TORSION ANGLE RESTRAINTS DERIVED USING UNLABELED, 15N-LABELED, AND 13C, 15N-LABELED SAMPLES OF THE LEADZYME PREPARED BY IN VITRO TRANSCRIPTION. |
- Sample preparation
Sample preparation
| Details | Contents: H2O/D2O |
|---|---|
| Sample conditions | Ionic strength: 0.1 M / pH: 5.5 / Pressure: 1 atm / Temperature: 298 K |
| Crystal grow | *PLUS Method: other / Details: NMR |
-NMR measurement
| NMR spectrometer |
|
|---|
- Processing
Processing
| Software |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR software |
| ||||||||||||
| Refinement | Method: SIMULATED ANNEALING FROM RANDOM TORSION ANGLES / Software ordinal: 1 Details: A COMPLETE SET OF REFINEMENT MACROS, ALONG WITH ASSOCIATED FILES, IS AVAILABLE UPON REQUEST TO THE AUTHORS. | ||||||||||||
| NMR ensemble | Conformer selection criteria: NO NOE VIOLATIONS GREATER THAN 0.3 A, NO DIHEDRAL VIOLATIONS GREATER THAN 3 DEGREES, GOOD STEREOCHEMICAL QUALITY, TOTAL ENERGY LESS THAN -120 KCAL/MOL, NOE PSEUDOENERGY LESS THAN 4 KCAL/ MOL Conformers calculated total number: 25 / Conformers submitted total number: 25 |
 Movie
Movie Controller
Controller








 PDBj
PDBj





























 HSQC
HSQC