+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1lbu | ||||||
|---|---|---|---|---|---|---|---|
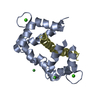
| タイトル | HYDROLASE METALLO (ZN) DD-PEPTIDASE | ||||||
 要素 要素 | MURAMOYL-PENTAPEPTIDE CARBOXYPEPTIDASE | ||||||
 キーワード キーワード | HYDROLASE / NUCLEAR RECEPTOR / CARBOXYPEPTIDASE | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報zinc D-Ala-D-Ala carboxypeptidase / zinc D-Ala-D-Ala carboxypeptidase activity / cell wall organization / proteolysis / extracellular region / metal ion binding 類似検索 - 分子機能 | ||||||
| 生物種 |  Streptomyces albus (バクテリア) Streptomyces albus (バクテリア) | ||||||
| 手法 |  X線回折 / 解像度: 1.8 Å X線回折 / 解像度: 1.8 Å | ||||||
 データ登録者 データ登録者 | Charlier, P. / Wery, J.-P. / Dideberg, O. / Frere, J.-M. | ||||||
 引用 引用 | ジャーナル: Handbook of Metalloproteins / 年: 2004 タイトル: Streptomyces Albus G D-Ala-A-Ala Carboxypeptidase 著者: Charlier, P. / Wery, J.-P. / Dideberg, O. / Frere, J.-M. #1:  ジャーナル: FEBS Lett. / 年: 1994 ジャーナル: FEBS Lett. / 年: 1994タイトル: Binding Site-Shaped Repeated Sequences of Bacterial Wall Peptidoglycan Hydrolases 著者: Ghuysen, J.M. / Lamotte-Brasseur, J. / Joris, B. / Shockman, G.D. #2:  ジャーナル: Nature / 年: 1982 ジャーナル: Nature / 年: 1982タイトル: Structure of a Zn2+-Containing D-Alanyl-D-Alanine-Cleaving Carboxypeptidase at 2.5 A Resolution 著者: Dideberg, O. / Charlier, P. / Dive, G. / Joris, B. / Frere, J.M. / Ghuysen, J.M. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1lbu.cif.gz 1lbu.cif.gz | 56.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1lbu.ent.gz pdb1lbu.ent.gz | 40.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1lbu.json.gz 1lbu.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1lbu_validation.pdf.gz 1lbu_validation.pdf.gz | 365 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1lbu_full_validation.pdf.gz 1lbu_full_validation.pdf.gz | 371.6 KB | 表示 | |
| XML形式データ |  1lbu_validation.xml.gz 1lbu_validation.xml.gz | 6.5 KB | 表示 | |
| CIF形式データ |  1lbu_validation.cif.gz 1lbu_validation.cif.gz | 10.5 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/lb/1lbu https://data.pdbj.org/pub/pdb/validation_reports/lb/1lbu ftp://data.pdbj.org/pub/pdb/validation_reports/lb/1lbu ftp://data.pdbj.org/pub/pdb/validation_reports/lb/1lbu | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 22184.465 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Streptomyces albus (バクテリア) / 株: G / 参照: UniProt: P00733, EC: 3.4.17.8 Streptomyces albus (バクテリア) / 株: G / 参照: UniProt: P00733, EC: 3.4.17.8 |
|---|---|
| #2: 化合物 | ChemComp-ZN / |
| #3: 水 | ChemComp-HOH / |
| 構成要素の詳細 | MURAMOYL-PENTAPEPTIDE CARBOXYPEPTIDASE HYDROLYSES INSOLUBLE PEPTIDOGLYCAN CLEAVAGE OF UDP-N- ...MURAMOYL-PENTAPEPTI |
| Has protein modification | Y |
| 配列の詳細 | THE FIRST AMINO ACID OF THE NATIVE PROTEIN IS ASP 1 WHICH IS PARTIALLY BLOCKED IN THIS STRUCTURE AS ...THE FIRST AMINO ACID OF THE NATIVE PROTEIN IS ASP 1 WHICH IS PARTIALLY BLOCKED IN THIS STRUCTURE AS A RESULT OF THE CYCLIZATIO |
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.17 Å3/Da / 溶媒含有率: 43.39 % |
|---|
-データ収集
| 放射 | 散乱光タイプ: x-ray |
|---|---|
| 放射波長 | 相対比: 1 |
| 反射 | 解像度: 1.8→30 Å / Num. obs: 15460 / % possible obs: 100 % / Observed criterion σ(I): 0 |
- 解析
解析
| ソフトウェア | 名称: PROLSQ / 分類: 精密化 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 1.8→6 Å / σ(F): 2 詳細: AMINO ACIDS 43 - 109 ARE SIMILAR TO AMINO ACIDS 194 - 261 OF B. SUBTILIS AUTOLYSIN.
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.8→6 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj




