+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1l0q | ||||||
|---|---|---|---|---|---|---|---|
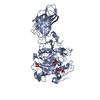
| タイトル | Tandem YVTN beta-propeller and PKD domains from an archaeal surface layer protein | ||||||
 要素 要素 | Surface layer protein | ||||||
 キーワード キーワード | PROTEIN BINDING / surface layer protein / SLP / S-layer / 7-bladed beta-propeller / PKD superfamily | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  Methanosarcina mazei (古細菌) Methanosarcina mazei (古細菌) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  多波長異常分散 / 解像度: 2.4 Å 多波長異常分散 / 解像度: 2.4 Å | ||||||
 データ登録者 データ登録者 | Jing, H. / Takagi, J. / Liu, J.-H. / Lindgren, S. / Zhang, R.-G. / Joachimiak, A. / Wang, J.-H. / Springer, T.A. | ||||||
 引用 引用 |  ジャーナル: Structure / 年: 2002 ジャーナル: Structure / 年: 2002タイトル: Archaeal Surface Layer Proteins Contain beta Propeller, PKD, and beta Helix Domains and Are Related to Metazoan Cell Surface Proteins. 著者: Jing, H. / Takagi, J. / Liu, J. / Lindgren, S. / Zhang, R. / Joachimiak, A. / Wang, J. / Springer, T. #1:  ジャーナル: Nat.Struct.Biol. / 年: 2001 ジャーナル: Nat.Struct.Biol. / 年: 2001タイトル: Implications for familial hypercholesterolemia from structure of the LDL receptor YWTD-EGF doman pair 著者: Jeon, H. / Meng, W. / Takagi, J. / Eck, M.J. / Springer, T.A. / Blacklow, S.C. #2:  ジャーナル: Embo J. / 年: 1999 ジャーナル: Embo J. / 年: 1999タイトル: The structure of a PKD domain from polycystin-1: implications for polycystic kidney disease 著者: Bycroft, M. / Bateman, A. / Clarke, J. / Hamill, S.J. / Sandford, R. / Thomas, R.L. / Chothia, C. #3:  ジャーナル: Arch.Microbiol. / 年: 1998 ジャーナル: Arch.Microbiol. / 年: 1998タイトル: Structure, organization, and expression of genes coding for envelope components in the archaeon Methanosarcina mazei S-6 著者: Mayerhofer, L.E. / Conway de Macario, E. / Yao, R. / Macario, A.J.L. #4:  ジャーナル: To be Published ジャーナル: To be Publishedタイトル: The genome of Methanosarcina acetivorans reveals extensive metabolic and physiological diversity 著者: Galagan, J.E. / Nusbaum, C. / Roy, A. / Endrizzi, M.G. / Macdonald, P. / FitzHugh, W. / Calvo, S. / Engels, R. / Smirnov, S. #5:  ジャーナル: J.Mol.Biol. / 年: 1998 ジャーナル: J.Mol.Biol. / 年: 1998タイトル: An extracellular beta-propeller module predicted in lipoprotein and scavenger receptors, tyrosine kinases, epidermal growth factor precursor, and extracellular matrix components 著者: Springer, T.A. | ||||||
| 履歴 |
| ||||||
| Remark 300 | BIOMOLECULE: 1 THIS ENTRY CONTAINS THE CRYSTALLOGRAPHIC ASYMMETRIC UNIT WHICH CONSISTS OF 4 ... BIOMOLECULE: 1 THIS ENTRY CONTAINS THE CRYSTALLOGRAPHIC ASYMMETRIC UNIT WHICH CONSISTS OF 4 CHAIN(S). SEE REMARK 350 FOR INFORMATION ON GENERATING THE BIOLOGICAL MOLECULE(S). THE WHOLE SURFACE LAYER PROTEIN MOLECULE HAS A DOMAIN ORGANIZATION OF YVTN-PKD-YVTN-(PKD)11. THE COORDINATES OF ANY ONE OF THE 4 MOLECULES IN THE ASYMMETRIC UNIT COULD CORRESPOND TO THE BIOLOGICAL CONFORMATION OF THE N-TERMINAL YVTN-PKD DOMAIN PAIR. |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1l0q.cif.gz 1l0q.cif.gz | 318.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1l0q.ent.gz pdb1l0q.ent.gz | 258.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1l0q.json.gz 1l0q.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1l0q_validation.pdf.gz 1l0q_validation.pdf.gz | 453.4 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1l0q_full_validation.pdf.gz 1l0q_full_validation.pdf.gz | 480.1 KB | 表示 | |
| XML形式データ |  1l0q_validation.xml.gz 1l0q_validation.xml.gz | 82.4 KB | 表示 | |
| CIF形式データ |  1l0q_validation.cif.gz 1l0q_validation.cif.gz | 112.5 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/l0/1l0q https://data.pdbj.org/pub/pdb/validation_reports/l0/1l0q ftp://data.pdbj.org/pub/pdb/validation_reports/l0/1l0q ftp://data.pdbj.org/pub/pdb/validation_reports/l0/1l0q | HTTPS FTP |
-関連構造データ
| 関連構造データ | |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 40796.559 Da / 分子数: 4 / 断片: YVTN beta-propeller domain 1 and PKD domain 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Methanosarcina mazei (古細菌) / 株: S-6 / プラスミド: pQE-60 / 発現宿主: Methanosarcina mazei (古細菌) / 株: S-6 / プラスミド: pQE-60 / 発現宿主:  #2: 水 | ChemComp-HOH / | Has protein modification | Y | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.16 Å3/Da / 溶媒含有率: 42.7 % | |||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 温度: 298 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 3.8 詳細: PEG-8000, potassium chloride, sodium acetate, pH 3.8, VAPOR DIFFUSION, HANGING DROP, temperature 298.0K | |||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS | |||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 200 K | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 19-ID / 波長: 0.979067,0.979221,0.939270 / ビームライン: 19-ID / 波長: 0.979067,0.979221,0.939270 | ||||||||||||
| 検出器 | タイプ: SBC-2 / 検出器: CCD / 日付: 1999年11月24日 / 詳細: mirrors | ||||||||||||
| 放射 | モノクロメーター: SAGITALLY FOCUSED Si(111) / プロトコル: MAD / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | ||||||||||||
| 放射波長 |
| ||||||||||||
| 反射 | 解像度: 2.4→100 Å / Num. all: 52881 / Num. obs: 52881 / % possible obs: 93.7 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / 冗長度: 6 % / Biso Wilson estimate: 30.36 Å2 / Rmerge(I) obs: 0.109 / Net I/σ(I): 14.6 | ||||||||||||
| 反射 シェル | 解像度: 2.4→2.49 Å / Rmerge(I) obs: 0.434 / Mean I/σ(I) obs: 3.1 / Num. unique all: 4965 / % possible all: 89 | ||||||||||||
| 反射 | *PLUS 最低解像度: 100 Å / Num. measured all: 318719 / Rmerge(I) obs: 0.109 | ||||||||||||
| 反射 シェル | *PLUS % possible obs: 89 % / Rmerge(I) obs: 0.434 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  多波長異常分散 / 解像度: 2.4→20 Å / Data cutoff high rms absF: 1000 / Isotropic thermal model: isotropic / 交差検証法: THROUGHOUT / σ(F): 0 / 立体化学のターゲット値: Engh & Huber 多波長異常分散 / 解像度: 2.4→20 Å / Data cutoff high rms absF: 1000 / Isotropic thermal model: isotropic / 交差検証法: THROUGHOUT / σ(F): 0 / 立体化学のターゲット値: Engh & Huber
| ||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | Bsol: 40.168 Å2 / ksol: 0.323 e/Å3 | ||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 23.4 Å2 | ||||||||||||||||||||||||||||||||||||
| Refine analyze |
| ||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.4→20 Å
| ||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.4→2.49 Å / Total num. of bins used: 10
| ||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS 最低解像度: 20 Å / % reflection Rfree: 5 % / Rfactor obs: 0.217 / Rfactor Rfree: 0.263 / Rfactor Rwork: 0.214 | ||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | ||||||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
| ||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | *PLUS Rfactor Rfree: 0.3466 / Rfactor Rwork: 0.2897 |
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj
