[English] 日本語
 Yorodumi
Yorodumi- PDB-1k4h: CRYSTAL STRUCTURE OF TRNA-GUANINE TRANSGLYCOSYLASE (TGT) COMPLEXE... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 1k4h | ||||||
|---|---|---|---|---|---|---|---|
| Title | CRYSTAL STRUCTURE OF TRNA-GUANINE TRANSGLYCOSYLASE (TGT) COMPLEXED WITH 2,6-Diamino-8-propylsulfanylmethyl-3H-quinazoline-4-one | ||||||
 Components Components | TRNA-GUANINE-TRANSGLYCOSYLASE | ||||||
 Keywords Keywords | TRANSFERASE / TRNA-MODIFYING ENZYME / GLYCOSYLTRANSFERASE | ||||||
| Function / homology |  Function and homology information Function and homology informationtRNA-guanosine34 preQ1 transglycosylase / tRNA-guanosine(34) queuine transglycosylase activity / tRNA queuosine(34) biosynthetic process / metal ion binding / cytosol Similarity search - Function | ||||||
| Biological species |  Zymomonas mobilis (bacteria) Zymomonas mobilis (bacteria) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  FOURIER SYNTHESIS / Resolution: 1.8 Å FOURIER SYNTHESIS / Resolution: 1.8 Å | ||||||
 Authors Authors | Brenk, R. / Meyer, E.A. / Castellano, R.K. / Furler, M. / Stubbs, M.T. / Klebe, G. / Diederich, F. | ||||||
 Citation Citation |  Journal: Chembiochem / Year: 2002 Journal: Chembiochem / Year: 2002Title: De novo design, synthesis, and in vitro evaluation of inhibitors for prokaryotic tRNA-guanine transglycosylase: a dramatic sulfur effect on binding affinity. Authors: Meyer, E.A. / Brenk, R. / Castellano, R.K. / Furler, M. / Klebe, G. / Diederich, F. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  1k4h.cif.gz 1k4h.cif.gz | 95.9 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb1k4h.ent.gz pdb1k4h.ent.gz | 70.8 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  1k4h.json.gz 1k4h.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/k4/1k4h https://data.pdbj.org/pub/pdb/validation_reports/k4/1k4h ftp://data.pdbj.org/pub/pdb/validation_reports/k4/1k4h ftp://data.pdbj.org/pub/pdb/validation_reports/k4/1k4h | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  1k4gC  1pudS S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| Unit cell |
|
- Components
Components
| #1: Protein | Mass: 42925.703 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Zymomonas mobilis (bacteria) / Production host: Zymomonas mobilis (bacteria) / Production host:  References: UniProt: P28720, tRNA-guanosine34 preQ1 transglycosylase |
|---|---|
| #2: Chemical | ChemComp-ZN / |
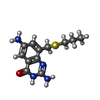
| #3: Chemical | ChemComp-APQ / |
| #4: Water | ChemComp-HOH / |
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.42 Å3/Da / Density % sol: 49.25 % | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Crystal grow | Temperature: 290 K / Method: 8.5 / pH: 8.5 / Details: TRIS, PEG 8000, DMSO, pH 8.50, temperature 290K | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Crystal grow | *PLUS Temperature: 25 ℃ / pH: 7.5 / Method: vapor diffusion, hanging dropDetails: Romier, C., (1996) Proteins: Struct.,Funct., Genet., 24, 516. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Components of the solutions | *PLUS
|
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  ROTATING ANODE / Type: RIGAKU RU300 / Wavelength: 1.54 ROTATING ANODE / Type: RIGAKU RU300 / Wavelength: 1.54 |
| Detector | Type: RIGAKU RAXIS IV / Detector: IMAGE PLATE / Date: Mar 5, 2001 / Details: mirrors |
| Radiation | Monochromator: YALE MIRRORS / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1.54 Å / Relative weight: 1 |
| Reflection | Resolution: 1.8→100 Å / Num. obs: 38143 / % possible obs: 99.6 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / Redundancy: 9.22 % / Rsym value: 0.05 / Net I/σ(I): 16.26 |
| Reflection shell | Resolution: 1.8→1.86 Å / Mean I/σ(I) obs: 3.96 / Rsym value: 0.247 / % possible all: 99.6 |
| Reflection | *PLUS Num. measured all: 351534 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  FOURIER SYNTHESIS FOURIER SYNTHESISStarting model: 1PUD Resolution: 1.8→40.8 Å / σ(F): 2 / Stereochemistry target values: Engh & Huber
| |||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 1.8→40.8 Å
| |||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||
| LS refinement shell | Resolution: 1.8→1.83 Å
| |||||||||||||||||||||||||
| Refinement | *PLUS Num. reflection obs: 38143 / Rfactor obs: 0.196 / Rfactor Rfree: 0.229 / Rfactor Rwork: 0.196 | |||||||||||||||||||||||||
| Solvent computation | *PLUS | |||||||||||||||||||||||||
| Displacement parameters | *PLUS | |||||||||||||||||||||||||
| LS refinement shell | *PLUS Rfactor Rfree: 0.35 / Rfactor Rwork: 0.318 / Rfactor obs: 0.318 |
 Movie
Movie Controller
Controller

















 PDBj
PDBj