+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1k46 | ||||||
|---|---|---|---|---|---|---|---|
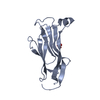
| タイトル | Crystal Structure of the Type III Secretory Domain of Yersinia YopH Reveals a Domain-Swapped Dimer | ||||||
 要素 要素 | PROTEIN-TYROSINE PHOSPHATASE YOPH | ||||||
 キーワード キーワード | HYDROLASE / domain-swap / phosphopeptide-binding domain / type III secretion domain | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報protein tyrosine phosphatase activity, metal-dependent / histone H2AXY142 phosphatase activity / non-membrane spanning protein tyrosine phosphatase activity / protein-tyrosine-phosphatase / extracellular region 類似検索 - 分子機能 | ||||||
| 生物種 |  Yersinia pseudotuberculosis (仮性結核菌) Yersinia pseudotuberculosis (仮性結核菌) | ||||||
| 手法 |  X線回折 / X線回折 /  多重同系置換 / 解像度: 2.2 Å 多重同系置換 / 解像度: 2.2 Å | ||||||
 データ登録者 データ登録者 | Smith, C.L. / Khandelwal, P. / Keliikuli, K. / Zuiderweg, E.R.P. / Saper, M.A. | ||||||
 引用 引用 |  ジャーナル: Mol.Microbiol. / 年: 2001 ジャーナル: Mol.Microbiol. / 年: 2001タイトル: Structure of the type III secretion and substrate-binding domain of Yersinia YopH phosphatase. 著者: Smith, C.L. / Khandelwal, P. / Keliikuli, K. / Zuiderweg, E.R. / Saper, M.A. #1:  ジャーナル: J.Biol.Chem. / 年: 2001 ジャーナル: J.Biol.Chem. / 年: 2001タイトル: Identification of Residues in the N-terminal Domain of the Yersinia Tyrosine Phosphatase that are Critical for Substrate Recognition 著者: Montagna, L.G. / Ivanov, M.I. / Bliska, J.B. #2:  ジャーナル: Mol.Microbiol. / 年: 1998 ジャーナル: Mol.Microbiol. / 年: 1998タイトル: Identification of an Amino-terminal Substrate-binding Domain in the Yersinia Tyrosine Phosphatase that is Required for Efficient Recognition of Focal Adhesion Targets 著者: Black, D.S. / Montagna, L.G. / Zitsmann, S. / Bliska, J.B. | ||||||
| 履歴 |
| ||||||
| Remark 300 | BIOMOLECULE: 1 THIS ENTRY CONTAINS THE CRYSTALLOGRAPHIC ASYMMETRIC UNIT WHICH CONSISTS OF 1 CHAIN(S) ...BIOMOLECULE: 1 THIS ENTRY CONTAINS THE CRYSTALLOGRAPHIC ASYMMETRIC UNIT WHICH CONSISTS OF 1 CHAIN(S). THIS IS LIKELY THE BIOLOGICALLY-RELEVANT STRUCTURE. THE RELEVANCE OF THE DOMAIN-SWAPPED DIMER OBSERVED IN THE CRYSTAL STRUCTURE IS DESCRIBED IN THE JRNL REFERENCE. ALSO SEE REMARK 900. THE OTHER MOLECULE OF THE DOMAIN-SWAPPED DIMER IS GENERATED BY APPLYING CRYSTALLOGRAPHIC OPERATOR 3 IN REMARK 290 FOLLOWED BY A TRANSLATION OF 47.90 IN X. |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1k46.cif.gz 1k46.cif.gz | 38.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1k46.ent.gz pdb1k46.ent.gz | 26.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1k46.json.gz 1k46.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1k46_validation.pdf.gz 1k46_validation.pdf.gz | 425 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1k46_full_validation.pdf.gz 1k46_full_validation.pdf.gz | 427.4 KB | 表示 | |
| XML形式データ |  1k46_validation.xml.gz 1k46_validation.xml.gz | 8.3 KB | 表示 | |
| CIF形式データ |  1k46_validation.cif.gz 1k46_validation.cif.gz | 10.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/k4/1k46 https://data.pdbj.org/pub/pdb/validation_reports/k4/1k46 ftp://data.pdbj.org/pub/pdb/validation_reports/k4/1k46 ftp://data.pdbj.org/pub/pdb/validation_reports/k4/1k46 | HTTPS FTP |
-関連構造データ
| 関連構造データ | |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
| ||||||||
| 詳細 | YopH(1-129) can exist as a monomer or dimer in solution. Crystal structure is a domain-swapped dimer consisting of two molecules related by a crystallographic two fold axis: -x+1,y,-z+1/2. The physiologically-relevant form is likely the monomer. |
- 要素
要素
| #1: タンパク質 | 分子量: 14882.599 Da / 分子数: 1 / 断片: amino-terminal domain (residues 1-129) / 由来タイプ: 組換発現 由来: (組換発現)  Yersinia pseudotuberculosis (仮性結核菌) Yersinia pseudotuberculosis (仮性結核菌)遺伝子: yopH / プラスミド: pT7-7 / 生物種 (発現宿主): Escherichia coli / 発現宿主:  |
|---|---|
| #2: 水 | ChemComp-HOH / |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.37 Å3/Da / 溶媒含有率: 48.09 % | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 温度: 295 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 8 詳細: PEG 8000, sodium chlolride, Tris , VAPOR DIFFUSION, HANGING DROP, temperature 295K | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 温度: 22 ℃ / pH: 7.2 / 手法: 蒸気拡散法 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 120 K |
|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: RIGAKU RUH3R / 波長: 1.5418 Å 回転陽極 / タイプ: RIGAKU RUH3R / 波長: 1.5418 Å |
| 検出器 | タイプ: RIGAKU RAXIS IV / 検出器: IMAGE PLATE / 日付: 1998年8月11日 / 詳細: mirrors |
| 放射 | モノクロメーター: Yale double focusing mirrors / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.5418 Å / 相対比: 1 |
| 反射 | 解像度: 2.2→22 Å / Num. all: 7503 / Num. obs: 7503 / % possible obs: 99.8 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / 冗長度: 6.2 % / Biso Wilson estimate: 23.3 Å2 / Rmerge(I) obs: 0.05 / Rsym value: 0.05 / Net I/σ(I): 20.8 |
| 反射 シェル | 解像度: 2.2→2.28 Å / 冗長度: 6.1 % / Rmerge(I) obs: 0.136 / Mean I/σ(I) obs: 16.2 / Num. unique all: 720 / % possible all: 99.9 |
| 反射 | *PLUS Num. measured all: 68285 / Rmerge(I) obs: 0.05 |
| 反射 シェル | *PLUS % possible obs: 99.9 % / Num. unique obs: 720 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  多重同系置換 多重同系置換開始モデル: de novo 解像度: 2.2→22 Å / Isotropic thermal model: isotropic / 交差検証法: R-free / σ(F): 0 / σ(I): 0 / 立体化学のターゲット値: Engh & Huber
| |||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 30.8 Å2 | |||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.2→22 Å
| |||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.2→2.25 Å /
| |||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: CNS / バージョン: 0.9 / 分類: refinement | |||||||||||||||||||||||||
| 精密化 | *PLUS 最低解像度: 22 Å / σ(F): 0 / Rfactor obs: 0.224 / Rfactor Rfree: 0.258 | |||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | |||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS Biso mean: 30.8 Å2 | |||||||||||||||||||||||||
| LS精密化 シェル | *PLUS Rfactor Rwork: 0.222 / Rfactor obs: 0.222 |
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj



