[English] 日本語
 Yorodumi
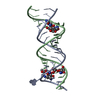
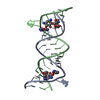
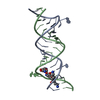
Yorodumi- PDB-1j7t: Complex between Paromomycin and the 16S-rRNA A-site at 2.5 A reso... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 1j7t | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Title | Complex between Paromomycin and the 16S-rRNA A-site at 2.5 A resolution | ||||||||||||||||||
 Components Components | 5'-R(* Keywords KeywordsRNA / RNA-aminoglycoside interactions / A site / UoU pairs / AA bulges | Function / homology | PAROMOMYCIN / RNA / RNA (> 10) |  Function and homology information Function and homology informationMethod |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.5 Å MOLECULAR REPLACEMENT / Resolution: 2.5 Å  Authors AuthorsVicens, Q. / Westhof, E. |  Citation Citation Journal: Structure / Year: 2001 Journal: Structure / Year: 2001Title: Crystal structure of paromomycin docked into the eubacterial ribosomal decoding A site. Authors: Vicens, Q. / Westhof, E. History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  1j7t.cif.gz 1j7t.cif.gz | 36.3 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb1j7t.ent.gz pdb1j7t.ent.gz | 25.7 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  1j7t.json.gz 1j7t.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/j7/1j7t https://data.pdbj.org/pub/pdb/validation_reports/j7/1j7t ftp://data.pdbj.org/pub/pdb/validation_reports/j7/1j7t ftp://data.pdbj.org/pub/pdb/validation_reports/j7/1j7t | HTTPS FTP |
|---|
-Related structure data
| Similar structure data |
|---|
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| Unit cell |
|
- Components
Components
| #1: RNA chain | Mass: 7048.259 Da / Num. of mol.: 2 / Source method: obtained synthetically / Details: Escherichia coli 16S rRNA A site #2: Chemical | #3: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.56 Å3/Da / Density % sol: 51.95 % | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Crystal grow | Temperature: 310 K / Method: vapor diffusion, hanging drop / pH: 6.4 Details: MPD, NaCl, MgSO4, glycerol, Na cacodylate, pH 6.4, VAPOR DIFFUSION, HANGING DROP, temperature 310K | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Components of the solutions |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Crystal grow | *PLUS Temperature: 21 ℃ | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Components of the solutions | *PLUS
|
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  ESRF ESRF  / Beamline: ID14-1 / Wavelength: 0.934 Å / Beamline: ID14-1 / Wavelength: 0.934 Å |
| Detector | Type: MARRESEARCH / Detector: CCD / Date: Sep 28, 2000 |
| Radiation | Monochromator: Diamond (111), Ge(220) / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.934 Å / Relative weight: 1 |
| Reflection | Resolution: 2.5→15 Å / Num. all: 5144 / Num. obs: 5144 / % possible obs: 95 % / Redundancy: 9.7 % / Rsym value: 0.055 / Net I/σ(I): 24.4 |
| Reflection shell | Resolution: 2.5→2.59 Å / Mean I/σ(I) obs: 6.5 / Rsym value: 0.23 / % possible all: 97.4 |
| Reflection | *PLUS Lowest resolution: 10 Å / Num. obs: 5278 / Rmerge(I) obs: 0.055 |
| Reflection shell | *PLUS % possible obs: 97.4 % / Rmerge(I) obs: 0.23 / Mean I/σ(I) obs: 6.4 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: Model containing the 16S rRNA A site as solved in the crystallographic structure of the 30S ribosomal particle complexed to paromomycin Resolution: 2.5→15 Å / Isotropic thermal model: isotropic / σ(F): 1.5 Stereochemistry target values: G. Parkinson, J. Vojtechovsky, L. Clowney, A.T. Brunger, H.M. Berman, New Parameters for the Refinement of Nucleic Acid Containing Structures, Acta Cryst. D, 52, 57-64 (1996)
| |||||||||||||||||||||||||
| Displacement parameters | Biso mean: 52.7 Å2 | |||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 2.5→15 Å
| |||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||
| Refinement | *PLUS Lowest resolution: 10 Å / % reflection Rfree: 7.5 % / Rfactor obs: 0.206 / Rfactor Rfree: 0.247 / Rfactor Rwork: 0.206 | |||||||||||||||||||||||||
| Solvent computation | *PLUS | |||||||||||||||||||||||||
| Displacement parameters | *PLUS | |||||||||||||||||||||||||
| Refine LS restraints | *PLUS
|
 Movie
Movie Controller
Controller




 PDBj
PDBj