+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1i25 | ||||||
|---|---|---|---|---|---|---|---|
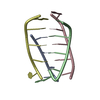
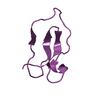
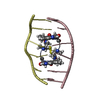
| タイトル | Three dimensional solution structure of huwentoxin-II by 2D 1H-NMR | ||||||
 要素 要素 | HUWENTOXIN-II | ||||||
 キーワード キーワード | TOXIN / neurotoxin / insecticidal toxin / disulfide bonds | ||||||
| 機能・相同性 | Huwentoxin-II family / Huwentoxin-II family / Huwentoxin-2 family signature. / host cell postsynaptic membrane / toxin activity / extracellular region / U1-theraphotoxin-Hs1a 機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  Ornithoctonus huwena (クモ) Ornithoctonus huwena (クモ) | ||||||
| 手法 | 溶液NMR / simulated annealing | ||||||
 データ登録者 データ登録者 | Shu, Q. / Lu, S.Y. / Gu, X.C. / Liang, S.P. | ||||||
 引用 引用 |  ジャーナル: Protein Sci. / 年: 2002 ジャーナル: Protein Sci. / 年: 2002タイトル: The structure of spider toxin huwentoxin-II with unique disulfide linkage: evidence for structural evolution. 著者: Shu, Q. / Lu, S.Y. / Gu, X.C. / Liang, S.P. #1:  ジャーナル: ACTA BIOCHIM.BIOPHYS.SINICA / 年: 2001 ジャーナル: ACTA BIOCHIM.BIOPHYS.SINICA / 年: 2001タイトル: Sequence-specific Assignment of 1H-NMR Resonance and Determination of the Secondary Structure of HWTX-II 著者: Shu, Q. / Lu, S.Y. / Gu, X.C. / Liang, S.P. #2:  ジャーナル: To be Published ジャーナル: To be Publishedタイトル: Assignment of Disulfide Bonds of Huwentoxin-II by Edman Degradation Sequencing and Stepwise Thiol Modification 著者: Shu, Q. / Huang, R.H. / Liang, S.P. #3:  ジャーナル: J.PEPT.RES. / 年: 1999 ジャーナル: J.PEPT.RES. / 年: 1999タイトル: Purification and Characterization of Huwentoxin-II, a Neurotoxic Peptide from the Venom of the Chinese Bird Spider Selenocosmia huwena 著者: Shu, Q. / Liang, S.P. | ||||||
| 履歴 |
| ||||||
| Remark 750 | TURN DETERMINATION METHOD: AUTHOR-DETERMINED |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1i25.cif.gz 1i25.cif.gz | 120.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1i25.ent.gz pdb1i25.ent.gz | 98.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1i25.json.gz 1i25.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1i25_validation.pdf.gz 1i25_validation.pdf.gz | 337.5 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1i25_full_validation.pdf.gz 1i25_full_validation.pdf.gz | 378 KB | 表示 | |
| XML形式データ |  1i25_validation.xml.gz 1i25_validation.xml.gz | 6.9 KB | 表示 | |
| CIF形式データ |  1i25_validation.cif.gz 1i25_validation.cif.gz | 10.5 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/i2/1i25 https://data.pdbj.org/pub/pdb/validation_reports/i2/1i25 ftp://data.pdbj.org/pub/pdb/validation_reports/i2/1i25 ftp://data.pdbj.org/pub/pdb/validation_reports/i2/1i25 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質・ペプチド | 分子量: 4300.246 Da / 分子数: 1 / 由来タイプ: 天然 / 詳細: VENOM / 由来: (天然)  Ornithoctonus huwena (クモ) / 器官: VENOM GLAND / 参照: UniProt: P82959 Ornithoctonus huwena (クモ) / 器官: VENOM GLAND / 参照: UniProt: P82959 |
|---|---|
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
| ||||||||||||||||||||||||||||||||||||
| NMR実験の詳細 | Text: This structure was determined using standard 2D homonuclear techniques. |
- 試料調製
試料調製
| 詳細 |
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料状態 |
| |||||||||||||||
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
-NMR測定
| NMRスペクトロメーター | タイプ: Bruker DMX / 製造業者: Bruker / モデル: DMX / 磁場強度: 500 MHz |
|---|
- 解析
解析
| NMR software |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: simulated annealing / ソフトェア番号: 1 詳細: The structures are based on 592 NOE-derived distance constraints, 16 dihedral angel restraints and 9 fake distance restraints from disulfide bonds. | ||||||||||||
| 代表構造 | 選択基準: closest to the average | ||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: structures with the lowest energy 計算したコンフォーマーの数: 50 / 登録したコンフォーマーの数: 10 |
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj X-PLOR
X-PLOR