+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1hvo | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | ZINC-AND SEQUENCE-DEPENDENT BINDING TO NUCLEIC ACIDS BY THE N-TERMINAL ZINC FINGER DOMAIN OF THE HIV-1 NUCLEOCAPSID PROTEIN: NMR STRUCTURE OF THE COMPLEX WITH THE PSI-SITE ANALOG, D/ACGCC | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | Viral protein/DNA / DNA / ZINC FINGER DOMAIN / HIV-1 NUCLEOCAPSID PROTEIN / PSI-SITE ANALOG / Viral protein-DNA COMPLEX | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral budding via host ESCRT complex / host multivesicular body / viral nucleocapsid / viral translational frameshifting / host cell nucleus / host cell plasma membrane / virion membrane / structural molecule activity / RNA binding / zinc ion binding / membrane 類似検索 - 分子機能 | ||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) | ||||||
| 手法 | 溶液NMR / simulated annealing | ||||||
 データ登録者 データ登録者 | South, T.L. / Summers, M.F. | ||||||
 引用 引用 |  ジャーナル: Protein Sci. / 年: 1993 ジャーナル: Protein Sci. / 年: 1993タイトル: Zinc- and sequence-dependent binding to nucleic acids by the N-terminal zinc finger of the HIV-1 nucleocapsid protein: NMR structure of the complex with the Psi-site analog, dACGCC. 著者: South, T.L. / Summers, M.F. #1:  ジャーナル: Protein Sci. / 年: 1992 ジャーナル: Protein Sci. / 年: 1992タイトル: Nucleocapsid Zinc Fingers Detected in Retroviruses: Exafs Studies of Intact Viruses and the Solution-State Structure of the Nucleocapsid Protein from HIV-1 著者: Summers, M.F. / Henderson, L.E. / Chance, M.R. / Bess Junior, J.W. / South, T.L. / Blake, P.R. / Sagi, I. / Perez-Alvarado, G. / Sowder III, R.C. / Hare, D.R. / Arthur, L.O. #2:  ジャーナル: Biochemistry / 年: 1991 ジャーナル: Biochemistry / 年: 1991タイトル: The C-Terminal Retroviral-Type Zinc Finger Domain from the HIV-1 Nucleocapsid Protein is Structurally Similar to the N-Terminal Zinc Finger Domain 著者: South, T.L. / Blake, P.R. / Hare, D.R. / Summers, M.F. #3:  ジャーナル: Biochemistry / 年: 1990 ジャーナル: Biochemistry / 年: 1990タイトル: The Nucleocapsid Protein Isolated from HIV-I Particles Binds Zinc and Form Retroviral-Type Zinc Fingers 著者: South, T.L. / Blake, P.R. / Sowder, R. / Arthur, L.O. / Henderson, L.E. / Summers, M.F. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1hvo.cif.gz 1hvo.cif.gz | 134 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1hvo.ent.gz pdb1hvo.ent.gz | 108.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1hvo.json.gz 1hvo.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1hvo_validation.pdf.gz 1hvo_validation.pdf.gz | 357.4 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1hvo_full_validation.pdf.gz 1hvo_full_validation.pdf.gz | 493.7 KB | 表示 | |
| XML形式データ |  1hvo_validation.xml.gz 1hvo_validation.xml.gz | 12.5 KB | 表示 | |
| CIF形式データ |  1hvo_validation.cif.gz 1hvo_validation.cif.gz | 20.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/hv/1hvo https://data.pdbj.org/pub/pdb/validation_reports/hv/1hvo ftp://data.pdbj.org/pub/pdb/validation_reports/hv/1hvo ftp://data.pdbj.org/pub/pdb/validation_reports/hv/1hvo | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 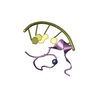
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: DNA鎖 | 分子量: 1465.001 Da / 分子数: 1 / 由来タイプ: 合成 /  キーワード: DEOXYRIBONUCLEIC ACID キーワード: DEOXYRIBONUCLEIC ACID |
|---|---|
| #2: タンパク質・ペプチド | 分子量: 2011.379 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)属: Lentivirus /  キーワード: POLYPEPTIDE / 参照: UniProt: Q70622 キーワード: POLYPEPTIDE / 参照: UniProt: Q70622 |
| #3: 化合物 | ChemComp-ZN / |
-実験情報
-実験
| 実験 | 手法: 溶液NMR |
|---|---|
| NMR実験の詳細 | Text: THE AVERAGE PAIRWISE RMDS'S FOR THE 40 DG/SA STRUCTURES (C,CA,N ATOMS OF RESIDUES A1-L51) IS 0.42 (0.07) ANGSTROMS. THE AVERAGE RMS DEVIATION FOR THE 40 DG/SA STRUCTURES RELATIVE TO THE MEAN ...Text: THE AVERAGE PAIRWISE RMDS'S FOR THE 40 DG/SA STRUCTURES (C,CA,N ATOMS OF RESIDUES A1-L51) IS 0.42 (0.07) ANGSTROMS. THE AVERAGE RMS DEVIATION FOR THE 40 DG/SA STRUCTURES RELATIVE TO THE MEAN COORDINATES (C,CA,N ATOMS OF RESIDUES A1-L51) IS 0.29 (0.07) ANGSTROMS. |
- 試料調製
試料調製
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
|---|
- 解析
解析
| NMR software | 名称: DSPACE / 分類: 精密化 |
|---|---|
| 精密化 | 手法: simulated annealing / ソフトェア番号: 1 詳細: THE INTERNAL CONFORMATION OF THE OLIGODEOXYRIBONUCLEOTIDE, D(ACGCC), WAS HELD FIXED IN AN IDEALIZED A-HELICAL CONFORMATION. THE A-HELIX STRUCTURE WAS GENERATED WITH QUANTA. SIMULATED ...詳細: THE INTERNAL CONFORMATION OF THE OLIGODEOXYRIBONUCLEOTIDE, D(ACGCC), WAS HELD FIXED IN AN IDEALIZED A-HELICAL CONFORMATION. THE A-HELIX STRUCTURE WAS GENERATED WITH QUANTA. SIMULATED ANNEALING WAS PERFORMED IN A MANNER THAT ALLOWED ATOMS OF THE ZINC FINGER ONLY TO MOVE DURING REFINEMENT. |
| NMRアンサンブル | 登録したコンフォーマーの数: 15 |
 ムービー
ムービー コントローラー
コントローラー







 PDBj
PDBj





