+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 1hhw | ||||||
|---|---|---|---|---|---|---|---|
| Title | Solution structure of LNA1:RNA hybrid | ||||||
 Components Components |
| ||||||
 Keywords Keywords | ANTISENSE / LOCKED NUCLEIC ACID / DNA / RNA / HYBRID / RNASE H | ||||||
| Function / homology | DNA/RNA hybrid / RNA Function and homology information Function and homology information | ||||||
| Method | SOLUTION NMR / simulated annealing | ||||||
 Authors Authors | Petersen, M. / Bondensgaard, K. / Wengel, J. / Jacobsen, J.P. | ||||||
 Citation Citation |  Journal: J.Am.Chem.Soc. / Year: 2002 Journal: J.Am.Chem.Soc. / Year: 2002Title: Locked Nucleic Acid (Lna) Recognition of RNA: NMR Solution Structures of Lna:RNA Hybrids Authors: Petersen, M. / Bondensgaard, K. / Wengel, J. / Jacobsen, J.P. #1:  Journal: Chemistry / Year: 2000 Journal: Chemistry / Year: 2000Title: Origin of Locked Nucleic Acid High-Affinity Recognition of RNA as Revealed by High-Resolution NMR Structures Authors: Petersen, M. / Bondensgaard, K. / Wengel, J. / Jacobsen, J.P. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  1hhw.cif.gz 1hhw.cif.gz | 454.5 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb1hhw.ent.gz pdb1hhw.ent.gz | 374.4 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  1hhw.json.gz 1hhw.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/hh/1hhw https://data.pdbj.org/pub/pdb/validation_reports/hh/1hhw ftp://data.pdbj.org/pub/pdb/validation_reports/hh/1hhw ftp://data.pdbj.org/pub/pdb/validation_reports/hh/1hhw | HTTPS FTP |
|---|
-Related structure data
- Links
Links
- Assembly
Assembly
| Deposited unit | 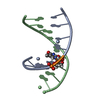
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR ensembles |
|
- Components
Components
| #1: DNA/RNA hybrid | Mass: 2758.821 Da / Num. of mol.: 1 / Source method: obtained synthetically |
|---|---|
| #2: RNA chain | Mass: 2855.767 Da / Num. of mol.: 1 / Source method: obtained synthetically |
| Sequence details | CHAIN A RESIDUE 5 IS A LOCKED NUCLEIC ACID OR (1R,3R,4R,7S) -7-HYDROXY-1-HYDROXYMETHYL-3-(THYMIN-1- ...CHAIN A RESIDUE 5 IS A LOCKED NUCLEIC ACID OR (1R,3R,4R,7S) -7-HYDROXY-1-HYDROXYMET |
-Experimental details
-Experiment
| Experiment | Method: SOLUTION NMR |
|---|---|
| NMR experiment | Type: NOESY |
- Sample preparation
Sample preparation
| Sample conditions | Ionic strength: 0.1 / pH: 7 / Pressure: 1 atm / Temperature: 298 K |
|---|---|
| Crystal grow | *PLUS Method: other / Details: NMR |
-NMR measurement
| NMR spectrometer | Type: Varian INOVA / Manufacturer: Varian / Model: INOVA / Field strength: 750 MHz |
|---|
- Processing
Processing
| NMR software |
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method: simulated annealing / Software ordinal: 1 Details: A TOTAL OF 284 NOE DISTANCE RESTRAINTS WERE OBTAINED FROM A FULL RELAXATION MATRIX ANALYSIS WITH THE PROGRAM RANDMARDI. 16 J-COUPLING CONSTANT RESTRAINTS WERE DERIVED FROM A J-SCALED-1H-31P- ...Details: A TOTAL OF 284 NOE DISTANCE RESTRAINTS WERE OBTAINED FROM A FULL RELAXATION MATRIX ANALYSIS WITH THE PROGRAM RANDMARDI. 16 J-COUPLING CONSTANT RESTRAINTS WERE DERIVED FROM A J-SCALED-1H-31P-HMBC SPECTRUM. 22 WATSON-CRICK BASE PAIRING RESTRAINTS. | |||||||||||||||
| NMR ensemble | Conformers calculated total number: 40 / Conformers submitted total number: 40 |
 Movie
Movie Controller
Controller







 PDBj
PDBj

 Amber
Amber