+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1gle | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | CATION PROMOTED ASSOCIATION (CPA) OF A REGULATORY AND TARGET PROTEIN IS CONTROLLED BY PHOSPHORYLATION | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | PHOSPHOTRANSFERASE | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of carbohydrate metabolic process / regulation of carbohydrate utilization / glycerol-3-phosphate metabolic process / negative regulation of maltose transport / enzyme IIA-maltose transporter complex / negative regulation of transmembrane transport / glycerol kinase / glycerol kinase activity / glycerol metabolic process / phosphoenolpyruvate-dependent sugar phosphotransferase system ...negative regulation of carbohydrate metabolic process / regulation of carbohydrate utilization / glycerol-3-phosphate metabolic process / negative regulation of maltose transport / enzyme IIA-maltose transporter complex / negative regulation of transmembrane transport / glycerol kinase / glycerol kinase activity / glycerol metabolic process / phosphoenolpyruvate-dependent sugar phosphotransferase system / transmembrane transporter complex / glycerol catabolic process / kinase activity / DNA damage response / zinc ion binding / ATP binding / metal ion binding / identical protein binding / membrane / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / 解像度: 2.94 Å X線回折 / 解像度: 2.94 Å | ||||||
 データ登録者 データ登録者 | Feese, M.D. / Meadow, N.D. / Roseman, S. / Pettigrew, D.W. / Remington, S.J. | ||||||
 引用 引用 |  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1994 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1994タイトル: Cation-promoted association of a regulatory and target protein is controlled by protein phosphorylation. 著者: Feese, M. / Pettigrew, D.W. / Meadow, N.D. / Roseman, S. / Remington, S.J. #1:  ジャーナル: Science / 年: 1993 ジャーナル: Science / 年: 1993タイトル: Three-Dimensional Structure of the Regulatory Complex of Escherichia Coli III-Glc with Glycerol Kinase 著者: Hurley, J.H. / Worthylake, D. / Faber, H.R. / Meadow, N.D. / Roseman, S. / Pettigrew, D.W. / Remington, S.J. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1gle.cif.gz 1gle.cif.gz | 139.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1gle.ent.gz pdb1gle.ent.gz | 105.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1gle.json.gz 1gle.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1gle_validation.pdf.gz 1gle_validation.pdf.gz | 476.8 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1gle_full_validation.pdf.gz 1gle_full_validation.pdf.gz | 545.2 KB | 表示 | |
| XML形式データ |  1gle_validation.xml.gz 1gle_validation.xml.gz | 23.5 KB | 表示 | |
| CIF形式データ |  1gle_validation.cif.gz 1gle_validation.cif.gz | 34.6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/gl/1gle https://data.pdbj.org/pub/pdb/validation_reports/gl/1gle ftp://data.pdbj.org/pub/pdb/validation_reports/gl/1gle ftp://data.pdbj.org/pub/pdb/validation_reports/gl/1gle | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 単位格子 |
| ||||||||
| Atom site foot note | 1: CIS PROLINE - PRO G 354 | ||||||||
| 詳細 | THE CRYSTAL CONTAINS TETRAMERS OF THE GLYCEROL KINASE/III-GLC COMPLEX WITH 222 POINT-GROUP SYMMETRY. THE TETRAMER IS LOCATED AT THE INTERSECTION OF THREE CRYSTALLOGRAPHIC TWO-FOLD AXES. THE FOLLOWING TRANSFORMATIONS WILL PRODUCE A TETRAMER OF THE COMPLEX WHEN APPLIED TO THE COORDINATES IN THIS ENTRY: TRANSFORM 1 (-X, 1-Y, Z) -1.0 0.0 0.0 0.0 0.0 -1.0 0.0 125.11 0.0 0.0 1.0 0.0 TRANSFORM 2 ( X, 1-Y, -Z) 1.0 0.0 0.0 0.0 0.0 -1.0 0.0 125.11 0.0 0.0 1.0 0.0 TRANSFORM 3 (-X, Y, -Z) -1.0 0.0 0.0 0.0 0.0 1.0 0.0 0.0 0.0 0.0 -1.0 0.0 |
- 要素
要素
-タンパク質 , 2種, 2分子 FG
| #1: タンパク質 | 分子量: 18141.834 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  参照: UniProt: P69783, protein-Npi-phosphohistidine-sugar phosphotransferase |
|---|---|
| #2: タンパク質 | 分子量: 56162.352 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  |
-非ポリマー , 4種, 52分子 
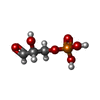





| #3: 化合物 | | #4: 化合物 | ChemComp-G3H / | #5: 化合物 | ChemComp-ADP / | #6: 水 | ChemComp-HOH / | |
|---|
-詳細
| 非ポリマーの詳細 | A ZN(II) ALSO BINDS AT THE ACTIVE SITE OF GLYCEROL KINASE BETWEEN THE PHOSPHORYL OXYGENS OF ADP AND ...A ZN(II) ALSO BINDS AT THE ACTIVE SITE OF GLYCEROL KINASE BETWEEN THE PHOSPHORYL |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.48 Å3/Da / 溶媒含有率: 64.65 % | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | *PLUS pH: 6.1 / 手法: 蒸気拡散法, ハンギングドロップ法 | ||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 放射 | 散乱光タイプ: x-ray |
|---|---|
| 放射波長 | 相対比: 1 |
- 解析
解析
| ソフトウェア | 名称: TNT / 分類: 精密化 | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 2.94→21.8 Å / Rfactor obs: 0.149 / σ(F): 0 | ||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.94→21.8 Å
| ||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: TNT / 分類: refinement | ||||||||||||||||||||||||||||||
| 精密化 | *PLUS Rfactor all: 0.149 | ||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | ||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj



