+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1fjl | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | HOMEODOMAIN FROM THE DROSOPHILA PAIRED PROTEIN BOUND TO A DNA OLIGONUCLEOTIDE | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | TRANSCRIPTION/DNA / DNA-BINDING PROTEIN / DNA / PAIRED BOX / TRANSCRIPTION REGULATION / TRANSCRIPTION-DNA COMPLEX | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報periodic partitioning by pair rule gene / HATs acetylate histones / RNA polymerase II transcription regulatory region sequence-specific DNA binding / nervous system development / DNA-binding transcription factor activity, RNA polymerase II-specific / RNA polymerase II cis-regulatory region sequence-specific DNA binding / regulation of transcription by RNA polymerase II / regulation of DNA-templated transcription / positive regulation of transcription by RNA polymerase II / DNA binding / nucleus 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / 解像度: 2 Å X線回折 / 解像度: 2 Å | ||||||
 データ登録者 データ登録者 | Wilson, D.S. / Guenther, B. / Desplan, C. / Kuriyan, J. | ||||||
 引用 引用 |  ジャーナル: Cell(Cambridge,Mass.) / 年: 1995 ジャーナル: Cell(Cambridge,Mass.) / 年: 1995タイトル: High resolution crystal structure of a paired (Pax) class cooperative homeodomain dimer on DNA. 著者: Wilson, D.S. / Guenther, B. / Desplan, C. / Kuriyan, J. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1fjl.cif.gz 1fjl.cif.gz | 86.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1fjl.ent.gz pdb1fjl.ent.gz | 61.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1fjl.json.gz 1fjl.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1fjl_validation.pdf.gz 1fjl_validation.pdf.gz | 461 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1fjl_full_validation.pdf.gz 1fjl_full_validation.pdf.gz | 485.1 KB | 表示 | |
| XML形式データ |  1fjl_validation.xml.gz 1fjl_validation.xml.gz | 16.1 KB | 表示 | |
| CIF形式データ |  1fjl_validation.cif.gz 1fjl_validation.cif.gz | 23.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/fj/1fjl https://data.pdbj.org/pub/pdb/validation_reports/fj/1fjl ftp://data.pdbj.org/pub/pdb/validation_reports/fj/1fjl ftp://data.pdbj.org/pub/pdb/validation_reports/fj/1fjl | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 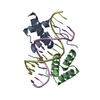
| ||||||||||
| 2 | 
| ||||||||||
| 単位格子 |
| ||||||||||
| 詳細 | A COMPLEX OF TWO HOMEODOMAINS (THE PROTEIN COMPONENT) BOUND TO ONE 14 BASE PAIR DNA DUPLEX IS THE MOLECULAR SPECIES ANALYZED IN THIS STUDY. EACH OF THE DNA DUPLEX AND THE COMPLEX HAS PSEUDO TWO-FOLD SYMMETRY ABOUT ITS CENTER. THE ASYMMETRIC UNIT CONTAINS 1.5 COMPLEXES, ONE OF WHICH IS COMPLETELY CONTAINED WITHIN THE ASYMMETRIC UNIT AND THE OTHER OF IS BISECTED AT ITS CENTER OF SYMMETRY BY A CRYSTALLOGRAPHIC TWO-FOLD AXIS. THE TWO-FOLD AVERAGED COMPLEX WAS MODELED AS A COMPLETE SINGLE HOMEODOMAIN AND A COMPLETE SINGLE STRAND OF THE DUPLEX. THEREFORE, THE MODEL CONTAINS THREE HOMEODOMAINS AND THREE DNA STRANDS. |
- 要素
要素
| #1: タンパク質 | 分子量: 9716.782 Da / 分子数: 3 / 断片: HOMEODOMAIN / 変異: C, C4S, S50Q / 由来タイプ: 組換発現 由来: (組換発現)  発現宿主:  #2: DNA鎖 | | 分子量: 4262.816 Da / 分子数: 1 / 由来タイプ: 合成 #3: DNA鎖 | | 分子量: 4293.827 Da / 分子数: 1 / 由来タイプ: 合成 #4: DNA鎖 | | 分子量: 4269.801 Da / 分子数: 1 / 由来タイプ: 合成 #5: 水 | ChemComp-HOH / | 配列の詳細 | THE STANDARD HOMEODOMAIN NUMBERING SYSTEM (SEE, FOR EXAMPLE, KISSINGER, C.R., LIU, B., MARTIN- ...THE STANDARD HOMEODOMAI | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.42 Å3/Da / 溶媒含有率: 49.09 % | |||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | *PLUS 温度: 4 ℃ / pH: 5.5 / 手法: unknown | |||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
|---|---|
| 放射波長 | 相対比: 1 |
| 反射 | *PLUS 最高解像度: 2 Å / 最低解像度: 20 Å / Num. obs: 28315 / % possible obs: 92.9 % / Num. measured all: 94960 / Rmerge(I) obs: 0.058 |
| 反射 シェル | *PLUS % possible obs: 83.3 % / Rmerge(I) obs: 0.185 |
- 解析
解析
| ソフトウェア | 名称:  X-PLOR / 分類: 精密化 X-PLOR / 分類: 精密化 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | Rfactor Rwork: 0.198 / Rfactor obs: 0.198 / 最高解像度: 2 Å 詳細: THE FOLLOWING SIDE CHAINS HAVE BEEN OMITTED FROM THE MODEL BECAUSE THEIR CONFORMATION WAS NOT EVIDENT FROM ELECTRON DENSITY MAPS: HOMEODOMAIN NO. 1: RESIDUES 122, 133, 158. HOMEODOMAIN NO. 2: ...詳細: THE FOLLOWING SIDE CHAINS HAVE BEEN OMITTED FROM THE MODEL BECAUSE THEIR CONFORMATION WAS NOT EVIDENT FROM ELECTRON DENSITY MAPS: HOMEODOMAIN NO. 1: RESIDUES 122, 133, 158. HOMEODOMAIN NO. 2: RESIDUES 233, 236, 239, 255, 258. HOMEODOMAIN NO. 3: RESIDUES 300, 324, 357, 358. DUE TO THE TWO-FOLD AVERAGING OF THE DNA DUPLEXES, ALTERNATE SIDE CHAIN IDENTITIES HAVE BEEN MODELED IN THE FOLLOWING WAY: ALTERNATE SIDE CHAIN FOR RESIDUE A D 1A: RESIDUE T D 1B ALTERNATE SIDE CHAIN FOR RESIDUE T D 8A: RESIDUE A D 8B ALTERNATE SIDE CHAIN FOR RESIDUE T E 1A: RESIDUE A E 1B ALTERNATE SIDE CHAIN FOR RESIDUE A E 8A: RESIDUE T E 8B ALTERNATE SIDE CHAIN FOR RESIDUE T F 1A: RESIDUE A F 1B ALTERNATE SIDE CHAIN FOR RESIDUE T F 8A: RESIDUE A F 8B TWO OF THE WATER MOLECULES ARE EXCLUDED BY ONE OF THE TWO ALTERNATE SIDE CHAINS AT DNA RESIDUES NO. 408 AND 508, AS FOLLOWS: WATER RESIDUE NO. 941 IS PRESENT WITH THE ALTERNATE SIDE CHAIN REPRESENTED BY RESIDUE 508 BUT NOT 528. WATER RESIDUE NO. 942 IS PRESENT WITH THE ALTERNATE SIDE CHAIN REPRESENTED BY RESIDUE 428 BUT NOT 408. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 最高解像度: 2 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS 最低解像度: 6 Å / Rfactor obs: 0.197 / Rfactor Rfree: 0.27 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS Biso mean: 38 Å2 |
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj







































