| 登録情報 | データベース: PDB / ID: 1f9s
|
|---|
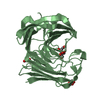
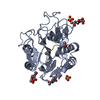
| タイトル | CRYSTAL STRUCTURE OF PLATELET FACTOR 4 MUTANT 2 |
|---|
 要素 要素 | PLATELET FACTOR 4 |
|---|
 キーワード キーワード | CYTOKINE / Platelet Factor 4 Mutant 2 |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
CXCR3 chemokine receptor binding / CXCR chemokine receptor binding / negative regulation of MHC class II biosynthetic process / killing by host of symbiont cells / positive regulation of macrophage differentiation / positive regulation of macrophage derived foam cell differentiation / leukocyte chemotaxis / chemokine activity / Chemokine receptors bind chemokines / defense response to protozoan ...CXCR3 chemokine receptor binding / CXCR chemokine receptor binding / negative regulation of MHC class II biosynthetic process / killing by host of symbiont cells / positive regulation of macrophage differentiation / positive regulation of macrophage derived foam cell differentiation / leukocyte chemotaxis / chemokine activity / Chemokine receptors bind chemokines / defense response to protozoan / monocyte chemotaxis / negative regulation of blood coagulation / negative regulation of extrinsic apoptotic signaling pathway in absence of ligand / Common Pathway of Fibrin Clot Formation / negative regulation of megakaryocyte differentiation / neutrophil chemotaxis / negative regulation of angiogenesis / platelet alpha granule lumen / Cell surface interactions at the vascular wall / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / platelet activation / adenylate cyclase-activating G protein-coupled receptor signaling pathway / cytokine-mediated signaling pathway / antimicrobial humoral immune response mediated by antimicrobial peptide / positive regulation of tumor necrosis factor production / Platelet degranulation / heparin binding / regulation of cell population proliferation / : / cellular response to lipopolysaccharide / G alpha (i) signalling events / inflammatory response / receptor ligand activity / positive regulation of gene expression / positive regulation of transcription by RNA polymerase II / extracellular space / extracellular region / cytoplasm類似検索 - 分子機能 CXC chemokine / CXC chemokine, conserved site / Small cytokines (intercrine/chemokine) C-x-C subfamily signature. / CXC Chemokine domain / Chemokine beta/gamma/delta / Intercrine alpha family (small cytokine C-X-C) (chemokine CXC). / Chemokine interleukin-8-like domain / Chemokine interleukin-8-like superfamily / Small cytokines (intecrine/chemokine), interleukin-8 like / OB fold (Dihydrolipoamide Acetyltransferase, E2P) - #40 ...CXC chemokine / CXC chemokine, conserved site / Small cytokines (intercrine/chemokine) C-x-C subfamily signature. / CXC Chemokine domain / Chemokine beta/gamma/delta / Intercrine alpha family (small cytokine C-X-C) (chemokine CXC). / Chemokine interleukin-8-like domain / Chemokine interleukin-8-like superfamily / Small cytokines (intecrine/chemokine), interleukin-8 like / OB fold (Dihydrolipoamide Acetyltransferase, E2P) - #40 / OB fold (Dihydrolipoamide Acetyltransferase, E2P) / Beta Barrel / Mainly Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / 解像度: 2.38 Å X線回折 / 解像度: 2.38 Å |
|---|
 データ登録者 データ登録者 | Yang, J. / Doyle, M. / Faulk, T. / Visentin, G. / Aster, R. / Edwards, B. |
|---|
 引用 引用 |  ジャーナル: To be Published ジャーナル: To be Published
タイトル: Structure Comparison of Two Platelet Factor 4 Mutants with the Wild-type Reveals the Epitopes for the Heparin-induced Thrombocytopenia Antibodies
著者: Yang, J. / Doyle, M. / Faulk, T. / Visentin, G. / Aster, R. / Edwards, B. |
|---|
| 履歴 | | 登録 | 2000年7月11日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2003年8月26日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年4月27日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2017年10月4日 | Group: Refinement description / カテゴリ: software |
|---|
| 改定 1.4 | 2021年11月3日 | Group: Database references / カテゴリ: database_2 / struct_ref_seq_dif
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_ref_seq_dif.details |
|---|
| 改定 1.5 | 2024年10月9日 | Group: Data collection / Structure summary
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / pdbx_entry_details / pdbx_modification_feature |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 / 解像度: 2.38 Å
X線回折 / 解像度: 2.38 Å  データ登録者
データ登録者 引用
引用 ジャーナル: To be Published
ジャーナル: To be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1f9s.cif.gz
1f9s.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1f9s.ent.gz
pdb1f9s.ent.gz PDB形式
PDB形式 1f9s.json.gz
1f9s.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 1f9s_validation.pdf.gz
1f9s_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 1f9s_full_validation.pdf.gz
1f9s_full_validation.pdf.gz 1f9s_validation.xml.gz
1f9s_validation.xml.gz 1f9s_validation.cif.gz
1f9s_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/f9/1f9s
https://data.pdbj.org/pub/pdb/validation_reports/f9/1f9s ftp://data.pdbj.org/pub/pdb/validation_reports/f9/1f9s
ftp://data.pdbj.org/pub/pdb/validation_reports/f9/1f9s リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / プラスミド: PT7-7 / 発現宿主:
Homo sapiens (ヒト) / プラスミド: PT7-7 / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 回転陽極 / タイプ: RIGAKU RU200 / 波長: 1.5418
回転陽極 / タイプ: RIGAKU RU200 / 波長: 1.5418  解析
解析 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj






