+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1dhi | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | LONG-RANGE STRUCTURAL EFFECTS IN A SECOND-SITE REVERTANT OF A MUTANT DIHYDROFOLATE REDUCTASE | ||||||
 要素 要素 | DIHYDROFOLATE REDUCTASE | ||||||
 キーワード キーワード | OXIDOREDUCTASE | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報methotrexate binding / dihydrofolic acid binding / 10-formyltetrahydrofolate biosynthetic process / response to methotrexate / NADP+ binding / folic acid biosynthetic process / folic acid binding / dihydrofolate metabolic process / dihydrofolate reductase / dihydrofolate reductase activity ...methotrexate binding / dihydrofolic acid binding / 10-formyltetrahydrofolate biosynthetic process / response to methotrexate / NADP+ binding / folic acid biosynthetic process / folic acid binding / dihydrofolate metabolic process / dihydrofolate reductase / dihydrofolate reductase activity / folic acid metabolic process / NADPH binding / tetrahydrofolate biosynthetic process / one-carbon metabolic process / NADP binding / response to xenobiotic stimulus / response to antibiotic / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / 解像度: 1.9 Å X線回折 / 解像度: 1.9 Å | ||||||
 データ登録者 データ登録者 | Oatley, S.J. / Villafranca, J.E. / Brown, K.A. / Kraut, J. | ||||||
 引用 引用 |  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1993 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1993タイトル: Long-range structural effects in a second-site revertant of a mutant dihydrofolate reductase. 著者: Brown, K.A. / Howell, E.E. / Kraut, J. #1:  ジャーナル: Biochemistry / 年: 1993 ジャーナル: Biochemistry / 年: 1993タイトル: How Do Mutation at Phenylalanine-153 and Isoleucine-155 Partially Suppress the Effects of the Aspartate-27-> Serine Mutation in Escherichia Coli Dihydrofolate Reductase 著者: Dion, A. / Linn, C.E. / Bradrick, T.D. / Georghiou, S. / Howell, E.E. #2:  ジャーナル: J.Biol.Chem. / 年: 1990 ジャーナル: J.Biol.Chem. / 年: 1990タイトル: Role of Aspartate 27 of Dihydrofolate Reductase from Escherichia Coli in Interconversion of Active and Inactive Enzyme Conformers and the Binding of Nadph 著者: Appleman, J.R. / Howell, E.E. / Kraut, J. / Blakely, R.L. #3:  ジャーナル: Biochemistry / 年: 1990 ジャーナル: Biochemistry / 年: 1990タイトル: A Second-Site Mutation at Phenylalanine-137 that Increases Catalytic Efficiency in the Mutant Aspartate-27-> Serine Escherichia Coli Dihydrofolate Reductase 著者: Howell, E.E. / Booth, C. / Farnum, M. / Kraut, J. / Warren, M.S. #4:  ジャーナル: Biochemistry / 年: 1990 ジャーナル: Biochemistry / 年: 1990タイトル: Dihydrofolate Reductase from Escherichia Coli: Probing the Role of Aspartate-27 and Phenylalanine-137 in Enzyme Conformation and the Binding of Nadph 著者: Dunn, S.M. / Lanigan, T.M. / Howell, E.E. #5:  ジャーナル: Science / 年: 1986 ジャーナル: Science / 年: 1986タイトル: Functional Role of Aspartic Acid-27 in Dihydrofolate Reductase Revealed by Mutagenesis 著者: Howell, E.E. / Villafranca, J.E. / Waren, M.S. / Oatley, S.J. / Kraut, J. #6:  ジャーナル: Biochemistry / 年: 1982 ジャーナル: Biochemistry / 年: 1982タイトル: Crystal Structures of Escherichia Coli and Lactobacillus Casei Dihydrofolate Reductase at 1.7 Angstroms Resolution. I. General Features and Binding of Methotrexate 著者: Bolin, J.T. / Filman, D.J. / Matthews, D.A. / Hamlin, R.C. / Kraut, J. #7:  ジャーナル: J.Biol.Chem. / 年: 1982 ジャーナル: J.Biol.Chem. / 年: 1982タイトル: Crystal Structures of Escherichia Coli and Lactobacillus Casei Dihydrofolate Reductase at 1.7 Angstroms Resolution. II. Environment of Bound Nadph and Implications for Catalysis 著者: Filman, D.J. / Bolin, J.T. / Matthews, D.A. / Kraut, J. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
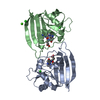
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1dhi.cif.gz 1dhi.cif.gz | 87.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1dhi.ent.gz pdb1dhi.ent.gz | 66.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1dhi.json.gz 1dhi.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1dhi_validation.pdf.gz 1dhi_validation.pdf.gz | 497.9 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1dhi_full_validation.pdf.gz 1dhi_full_validation.pdf.gz | 514.6 KB | 表示 | |
| XML形式データ |  1dhi_validation.xml.gz 1dhi_validation.xml.gz | 11.2 KB | 表示 | |
| CIF形式データ |  1dhi_validation.cif.gz 1dhi_validation.cif.gz | 17.4 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/dh/1dhi https://data.pdbj.org/pub/pdb/validation_reports/dh/1dhi ftp://data.pdbj.org/pub/pdb/validation_reports/dh/1dhi ftp://data.pdbj.org/pub/pdb/validation_reports/dh/1dhi | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
| ||||||||
| Atom site foot note | 1: GLY A 95 - GLY A 96 OMEGA = 2.83 PEPTIDE BOND DEVIATES SIGNIFICANTLY FROM TRANS CONFORMATION 2: GLY B 95 - GLY B 96 OMEGA = 2.85 PEPTIDE BOND DEVIATES SIGNIFICANTLY FROM TRANS CONFORMATION | ||||||||
| 非結晶学的対称性 (NCS) | NCS oper: (Code: given Matrix: (-0.9245, -0.37489, 0.06906), ベクター: 詳細 | THE TRANSFORMATION PRESENTED ON *MTRIX* RECORDS BELOW WILL YIELD APPROXIMATE COORDINATES FOR CHAIN *A* WHEN APPLIED TO CHAIN *B*. | |
- 要素
要素
| #1: タンパク質 | 分子量: 17992.316 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  #2: 化合物 | #3: 化合物 | #4: 化合物 | ChemComp-CA / | #5: 水 | ChemComp-HOH / | 配列の詳細 | SEQUENCE ADVISORY NOTICE: DIFFERENCE BETWEEN SWISS-PROT AND PDB SEQUENCE. SWISS-PROT ENTRY NAME: ...SEQUENCE ADVISORY NOTICE: DIFFERENCE | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.57 Å3/Da / 溶媒含有率: 52.2 % | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | *PLUS pH: 6.8 / 手法: unknown / 詳細: referred to J.Biol.Chem. 257.13650-13662 1982 | ||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 放射 | 散乱光タイプ: x-ray |
|---|---|
| 放射波長 | 相対比: 1 |
| 反射 | *PLUS 最高解像度: 1.9 Å / Num. obs: 26345 / % possible obs: 91 % / Rmerge(I) obs: 0.06 |
- 解析
解析
| ソフトウェア | 名称: TNT / 分類: 精密化 | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | Rfactor obs: 0.155 / 最高解像度: 1.9 Å | ||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 最高解像度: 1.9 Å
| ||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: TNT / 分類: refinement | ||||||||||||||||||||||||||||||
| 精密化 | *PLUS 最高解像度: 1.9 Å / Num. reflection obs: 26345 / Rfactor obs: 0.155 | ||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | ||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj