+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1dfn | ||||||
|---|---|---|---|---|---|---|---|
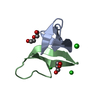
| タイトル | CRYSTAL STRUCTURE OF DEFENSIN HNP-3, AN AMPHIPHILIC DIMER: MECHANISMS OF MEMBRANE PERMEABILIZATION | ||||||
 要素 要素 | DEFENSIN HNP-3 | ||||||
 キーワード キーワード | DEFENSIN | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報pore-forming activity / disruption of plasma membrane integrity in another organism / Defensins / killing by host of symbiont cells / T cell chemotaxis / Alpha-defensins / defense response to protozoan / defense response to fungus / estrogen receptor signaling pathway / innate immune response in mucosa ...pore-forming activity / disruption of plasma membrane integrity in another organism / Defensins / killing by host of symbiont cells / T cell chemotaxis / Alpha-defensins / defense response to protozoan / defense response to fungus / estrogen receptor signaling pathway / innate immune response in mucosa / Golgi lumen / antimicrobial humoral immune response mediated by antimicrobial peptide / chemotaxis / azurophil granule lumen / antibacterial humoral response / cellular response to lipopolysaccharide / : / defense response to Gram-negative bacterium / defense response to virus / killing of cells of another organism / defense response to Gram-positive bacterium / immune response / Neutrophil degranulation / protein homodimerization activity / extracellular space / extracellular exosome / extracellular region 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / 解像度: 1.9 Å X線回折 / 解像度: 1.9 Å | ||||||
 データ登録者 データ登録者 | Hill, C.P. / Yee, J. / Selsted, M.E. / Eisenberg, D. | ||||||
 引用 引用 |  ジャーナル: Science / 年: 1991 ジャーナル: Science / 年: 1991タイトル: Crystal structure of defensin HNP-3, an amphiphilic dimer: mechanisms of membrane permeabilization. 著者: Hill, C.P. / Yee, J. / Selsted, M.E. / Eisenberg, D. #1:  ジャーナル: J.Clin.Invest. / 年: 1985 ジャーナル: J.Clin.Invest. / 年: 1985タイトル: Primary Structures of Three Human Neutrophil Defensins 著者: Selsted, M.E. / Harwig, S.S.L. / Ganz, T. / Schilling, J.W. / Lehrer, R.I. #2:  ジャーナル: J.Clin.Invest. / 年: 1985 ジャーナル: J.Clin.Invest. / 年: 1985タイトル: Defensins. Natural Peptide Antibiotics of Human Neutrophils 著者: Ganz, T. / Selsted, M.E. / Szklarek, D. / Harwig, S.S.L. / Daher, K. / Bainton, D.F. / Lehrer, R.I. | ||||||
| 履歴 |
| ||||||
| Remark 700 | SHEET THERE IS A CLASSIC BULGE INVOLVING RESIDUES 17, 18, AND 29 FOR EACH OF THE CHAINS. |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1dfn.cif.gz 1dfn.cif.gz | 23.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1dfn.ent.gz pdb1dfn.ent.gz | 15.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1dfn.json.gz 1dfn.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1dfn_validation.pdf.gz 1dfn_validation.pdf.gz | 422.4 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1dfn_full_validation.pdf.gz 1dfn_full_validation.pdf.gz | 424.9 KB | 表示 | |
| XML形式データ |  1dfn_validation.xml.gz 1dfn_validation.xml.gz | 5.3 KB | 表示 | |
| CIF形式データ |  1dfn_validation.cif.gz 1dfn_validation.cif.gz | 6.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/df/1dfn https://data.pdbj.org/pub/pdb/validation_reports/df/1dfn ftp://data.pdbj.org/pub/pdb/validation_reports/df/1dfn ftp://data.pdbj.org/pub/pdb/validation_reports/df/1dfn | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
| ||||||||
| Atom site foot note | 1: RESIDUES A 8 AND B 8 ARE CIS PROLINES. 2: THERE IS A CLASSIC BULGE INVOLVING RESIDUES 17, 18, AND 29 FOR EACH OF THE CHAINS. | ||||||||
| Components on special symmetry positions |
| ||||||||
| 詳細 | THE TWO MOLECULES IN THE ASYMMETRIC UNIT HAVE BEEN ASSIGNED CHAIN IDENTIFIERS *A* AND *B*. EACH OF THE TWO MONOMERS IN THE ASYMMETRIC UNIT CONTAINS A THREE STRAND ANTIPARALLEL SHEET. A LOCAL TWO-FOLD AXIS RELATING MOLECULES *A* AND *B* RESULTS IN A SIX-STRANDED ANTIPARALLEL SHEET IN THE DIMER. THE TWO MOLECULES IN THE ASYMMETRIC UNIT ARE RELATED TO EACH OTHER BY A LOCAL TWO-FOLD AXIS. |
- 要素
要素
| #1: タンパク質・ペプチド | 分子量: 3496.121 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 参照: UniProt: P59665, UniProt: P59666*PLUS Homo sapiens (ヒト) / 参照: UniProt: P59665, UniProt: P59666*PLUS#2: 水 | ChemComp-HOH / | Has protein modification | Y | 配列の詳細 | THE 30 RESIDUES IN HNP-3 HAVE BEEN NUMBERED FROM 2 - 31 IN ORDER TO BETTER FIT THE SEQUENCE ...THE 30 RESIDUES IN HNP-3 HAVE BEEN NUMBERED FROM 2 - 31 IN ORDER TO BETTER FIT THE SEQUENCE ALIGNMENT WITH OTHER DEFENSIN MOLECULES. | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2 Å3/Da / 溶媒含有率: 38.39 % | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | *PLUS pH: 4 / 手法: 蒸気拡散法, ハンギングドロップ法 | ||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 放射 | 散乱光タイプ: x-ray |
|---|---|
| 放射波長 | 相対比: 1 |
| 反射 | *PLUS 最高解像度: 1.9 Å / Num. obs: 4524 / % possible obs: 96.7 % / Observed criterion σ(F): 2 / Num. measured all: 22975 / Rmerge F obs: 0.054 / Biso Wilson estimate: 15.6 Å2 |
- 解析
解析
| ソフトウェア | 名称: PROLSQ / 分類: 精密化 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 1.9→10 Å / Rfactor obs: 0.19 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.9→10 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: PROLSQ / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS 最高解像度: 1.9 Å / 最低解像度: 10 Å / Num. reflection all: 4448 / Rfactor obs: 0.19 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj



