+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1d0u | ||||||
|---|---|---|---|---|---|---|---|
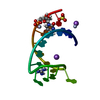
| タイトル | SOLUTION STRUCTURE OF AN RNA BINDING SITE FOR PHAGE MS2 COAT PROTEIN | ||||||
 要素 要素 | PHAGE MS2 RNA BINDING SITE | ||||||
 キーワード キーワード | RNA / RNA HAIRPIN / BULGED BASE / STEM-LOOP | ||||||
| 機能・相同性 | RNA / RNA (> 10) 機能・相同性情報 機能・相同性情報 | ||||||
| 手法 | 溶液NMR / simulated annealing, molecular dynamics refinement | ||||||
| Model type details | minimized average | ||||||
 データ登録者 データ登録者 | Smith, J.S. / Nikonowicz, E.P. | ||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 2000 ジャーナル: Biochemistry / 年: 2000タイトル: Phosphorothioate substitution can substantially alter RNA conformation. 著者: Smith, J.S. / Nikonowicz, E.P. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1d0u.cif.gz 1d0u.cif.gz | 302.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1d0u.ent.gz pdb1d0u.ent.gz | 253.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1d0u.json.gz 1d0u.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1d0u_validation.pdf.gz 1d0u_validation.pdf.gz | 323.4 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1d0u_full_validation.pdf.gz 1d0u_full_validation.pdf.gz | 466.8 KB | 表示 | |
| XML形式データ |  1d0u_validation.xml.gz 1d0u_validation.xml.gz | 12.8 KB | 表示 | |
| CIF形式データ |  1d0u_validation.cif.gz 1d0u_validation.cif.gz | 20.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/d0/1d0u https://data.pdbj.org/pub/pdb/validation_reports/d0/1d0u ftp://data.pdbj.org/pub/pdb/validation_reports/d0/1d0u ftp://data.pdbj.org/pub/pdb/validation_reports/d0/1d0u | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: RNA鎖 | 分子量: 6729.044 Da / 分子数: 1 / 由来タイプ: 合成 / 詳細: Transcription by T7 RNA ploymerase |
|---|
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
| ||||||||||||||||||||||||||||
| NMR実験の詳細 | Text: The structure was determined using standard 2D homonuclear and 2D/3D heteronuclear techniques |
- 試料調製
試料調製
| 詳細 |
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 試料状態 | イオン強度: ~50 mM / pH: 6.8 / 圧: ambient / 温度: 298 K | |||||||||
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
-NMR測定
| NMRスペクトロメーター | タイプ: Bruker AMX / 製造業者: Bruker / モデル: AMX / 磁場強度: 500 MHz |
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: simulated annealing, molecular dynamics refinement / ソフトェア番号: 1 詳細: structures were calculated using 469 conformationally restrictive NOE distance constraints (i.e., intra-residue sugar-sugar NOEs are not included), 20 H-bond base pair constraints, 30 ...詳細: structures were calculated using 469 conformationally restrictive NOE distance constraints (i.e., intra-residue sugar-sugar NOEs are not included), 20 H-bond base pair constraints, 30 backbone dihedral restraints, and dihedral restraints to restrict sugar pucker conformation. | ||||||||||||||||||||
| 代表構造 | 選択基準: minimized average structure | ||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: back calculated data agree with experimental NOESY spectrum,structures with acceptable covalent geometry,structures with the least restraint violations, ...コンフォーマー選択の基準: back calculated data agree with experimental NOESY spectrum,structures with acceptable covalent geometry,structures with the least restraint violations, structures with the lowest energy 計算したコンフォーマーの数: 111 / 登録したコンフォーマーの数: 23 |
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj





























 X-PLOR
X-PLOR