+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1chv | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | ELUCIDATION OF THE SOLUTION STRUCTURE OF CARDIOTOXIN ANALOGUE V FROM THE TAIWAN COBRA (NAJA NAJA ATRA) VENOM | ||||||
 要素 要素 | PROTEIN (CARDIOTOXIN ANALOGUE V) | ||||||
 キーワード キーワード | TOXIN / CARDIOTOXINS / CYTOTOXINS | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報other organism cell membrane / ion channel regulator activity / toxin activity / killing of cells of another organism / extracellular region / membrane 類似検索 - 分子機能 | ||||||
| 生物種 |  Naja atra (タイワンコブラ) Naja atra (タイワンコブラ) | ||||||
| 手法 | 溶液NMR / simulated annealing | ||||||
| Model type details | minimized average | ||||||
 データ登録者 データ登録者 | Jayaraman, G. / Kumar, T.K.S. / Tsai, C.C. / Yu, C. | ||||||
 引用 引用 |  ジャーナル: Protein Sci. / 年: 2000 ジャーナル: Protein Sci. / 年: 2000タイトル: Elucidation of the solution structure of cardiotoxin analogue V from the Taiwan cobra (Naja naja atra)--identification of structural features important for the lethal action of snake venom cardiotoxins 著者: Jayaraman, G. / Kumar, T.K.S. / Tsai, C.C. / Chou, S.H. / Ho, C.L. / Yu, C. #1: ジャーナル: Toxicon / 年: 1997 タイトル: Cardiotoxin-like basic protein (CLBP) from Naja naja atra is not a cardiotoxin. 著者: Sivaraman, T. / Kumar, T.K. / Yang, P.W. / Yu, C. #2: ジャーナル: J.Biomol.Struct.Dyn. / 年: 1997 タイトル: Snake venom cardiotoxins-structure, dynamics, function and folding. 著者: Kumar, T.K. / Jayaraman, G. / Lee, C.S. / Arunkumar, A.I. / Sivaraman, T. / Samuel, D. / Yu, C. #3: ジャーナル: Biochem.Biophys.Res.Commun. / 年: 1995 タイトル: Sequence comparison and computer modelling of cardiotoxins and cobrotoxin isolated from Taiwan cobra. 著者: Chiou, S.H. / Hung, C.C. / Huang, H.C. / Chen, S.T. / Wang, K.T. / Yang, C.C. #4: ジャーナル: Biochem.Int. / 年: 1985 タイトル: Amino acid sequence of a cardiotoxin-like basic polypeptide (CLBP) with low cytotoxic activity isolated from the venom of the Formosan cobra (Naja naja atra). 著者: Takechi, M. / Tanaka, Y. / Hayashi, K. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1chv.cif.gz 1chv.cif.gz | 31.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1chv.ent.gz pdb1chv.ent.gz | 22.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1chv.json.gz 1chv.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1chv_validation.pdf.gz 1chv_validation.pdf.gz | 345.2 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1chv_full_validation.pdf.gz 1chv_full_validation.pdf.gz | 349 KB | 表示 | |
| XML形式データ |  1chv_validation.xml.gz 1chv_validation.xml.gz | 3.4 KB | 表示 | |
| CIF形式データ |  1chv_validation.cif.gz 1chv_validation.cif.gz | 4.1 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ch/1chv https://data.pdbj.org/pub/pdb/validation_reports/ch/1chv ftp://data.pdbj.org/pub/pdb/validation_reports/ch/1chv ftp://data.pdbj.org/pub/pdb/validation_reports/ch/1chv | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 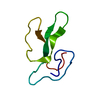
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 6821.410 Da / 分子数: 1 / 由来タイプ: 天然 詳細: CTX V OBTAINED FROM THE SNAKE (NAJA NAJA ATRA) VENOM 由来: (天然)  Naja atra (タイワンコブラ) / 参照: UniProt: P07525 Naja atra (タイワンコブラ) / 参照: UniProt: P07525 |
|---|---|
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
| ||||||||||||||||
| NMR実験の詳細 | Text: MEAN STRUCTURE. NULL |
- 試料調製
試料調製
| 詳細 | 内容: 90% H2O AND 10% D2O |
|---|---|
| 試料状態 | pH: 3 / 温度: 293 K |
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
-NMR測定
| NMRスペクトロメーター | タイプ: Bruker DMX600 / 製造業者: Bruker / モデル: DMX600 / 磁場強度: 600 MHz |
|---|
- 解析
解析
| NMR software |
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: simulated annealing / ソフトェア番号: 1 / 詳細: REFINE.INP | |||||||||
| 代表構造 | 選択基準: minimized average structure | |||||||||
| NMRアンサンブル | コンフォーマー選択の基準: LEAST RESTRAINT VIOLATION 計算したコンフォーマーの数: 50 / 登録したコンフォーマーの数: 1 |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj

 X-PLOR
X-PLOR