+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1chd | ||||||
|---|---|---|---|---|---|---|---|
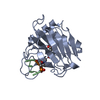
| タイトル | CHEB METHYLESTERASE DOMAIN | ||||||
 要素 要素 | CHEB METHYLESTERASE | ||||||
 キーワード キーワード | CARBOXYL METHYLESTERASE / CHEMOTAXIS PROTEIN / SERINE HYDROLASE | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報protein-glutamate methylesterase / protein-glutamate methylesterase activity / protein-glutamine glutaminase activity / protein-glutamine glutaminase / phosphorelay response regulator activity / chemotaxis / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  Salmonella typhimurium (サルモネラ菌) Salmonella typhimurium (サルモネラ菌) | ||||||
| 手法 |  X線回折 / 解像度: 1.75 Å X線回折 / 解像度: 1.75 Å | ||||||
 データ登録者 データ登録者 | West, A.H. / Martinez-Hackert, E. / Stock, A.M. | ||||||
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 1995 ジャーナル: J.Mol.Biol. / 年: 1995タイトル: Crystal structure of the catalytic domain of the chemotaxis receptor methylesterase, CheB. 著者: West, A.H. / Martinez-Hackert, E. / Stock, A.M. #1:  ジャーナル: To be Published ジャーナル: To be Publishedタイトル: Purification, Crystallization, and Preliminary X-Ray Diffraction Analyses of the Bacterial Chemotaxis Receptor Modifying Enzymes 著者: West, A.H. / Djordjevic, S. / Martinez-Hackert, E. / Stock, A.M. #2:  ジャーナル: Biochim.Biophys.Acta / 年: 1992 ジャーナル: Biochim.Biophys.Acta / 年: 1992タイトル: Evidence that the Methylesterase of Bacterial Chemotaxis May be a Serine Hydrolase 著者: Krueger, J.K. / Stock, J. / Schutt, C.E. #3:  ジャーナル: Annu.Rev.Biophys.Biophys.Chem. / 年: 1991 ジャーナル: Annu.Rev.Biophys.Biophys.Chem. / 年: 1991タイトル: Bacterial Chemotaxis and the Molecular Logic of Intracellular Signal Transduction Networks 著者: Stock, J.B. / Lukat, G.S. / Stock, A.M. #4:  ジャーナル: J.Biol.Chem. / 年: 1985 ジャーナル: J.Biol.Chem. / 年: 1985タイトル: Multiple Forms of the Cheb Methylesterase in Bacterial Chemosensing 著者: Simms, S.A. / Keane, M.G. / Stock, J. | ||||||
| 履歴 |
| ||||||
| Remark 650 | HELIX HELIX DETERMINATION METHOD: H BONDS, DIHEDRAL ANGLES, KABSCH AND SANDER ALGORITHM, AND VISUAL ...HELIX HELIX DETERMINATION METHOD: H BONDS, DIHEDRAL ANGLES, KABSCH AND SANDER ALGORITHM, AND VISUAL INSPECTION HELIX_ID: L1,PART OF CONNECTING LOOP BETWEEN BETA STRAND 2 AND HELIX B. HELIX_ID: L2,PART OF CONNECTING LOOP BETWEEN BETA STRAND 6 AND HELIX C. HELIX_ID: L3,PART OF CONNECTING LOOP BETWEEN BETA STRAND 9 AND HELIX F. HELIX_ID: L4,PART OF CONNECTING LOOP BETWEEN BETA STRAND 8 AND HELIX E. | ||||||
| Remark 700 | SHEET SHEET SHEET_ID: SH1, H BONDS, DIHEDRAL ANGLES, KABSCH AND SANDER ALGORITHM, AND VISUAL ...SHEET SHEET SHEET_ID: SH1, H BONDS, DIHEDRAL ANGLES, KABSCH AND SANDER ALGORITHM, AND VISUAL INSPECTION. CENTRAL PARALLEL BETA SHEET. SHEET_ID: SH2, ANTIPARALLEL BETA MOTIF THAT FLANKS ONE SIDE OF THE CENTRAL SEVEN-STRANDED BETA SHEET. |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1chd.cif.gz 1chd.cif.gz | 50.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1chd.ent.gz pdb1chd.ent.gz | 36.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1chd.json.gz 1chd.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ch/1chd https://data.pdbj.org/pub/pdb/validation_reports/ch/1chd ftp://data.pdbj.org/pub/pdb/validation_reports/ch/1chd ftp://data.pdbj.org/pub/pdb/validation_reports/ch/1chd | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
| ||||||||
| Atom site foot note | 1: CIS PROLINE - PRO 258 |
- 要素
要素
| #1: タンパク質 | 分子量: 21457.723 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Salmonella typhimurium (サルモネラ菌) Salmonella typhimurium (サルモネラ菌)株: LT2 / 遺伝子: CHEB / プラスミド: PCP1 (PUC12 VECTOR) / 遺伝子 (発現宿主): CHEB / 発現宿主:  | ||
|---|---|---|---|
| #2: 水 | ChemComp-HOH / | ||
| 構成要素の詳細 | TURN TURN TYPES CLASSIFIED| 由来についての詳細 | MOLECULE_NAME: CHEB METHYLESTERASE DOMAIN. DETAILS OF CONSTRUCTION OF EXPRESSION PLASMID AND ...MOLECULE_NAME: CHEB METHYLESTE | |
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.35 Å3/Da / 溶媒含有率: 47.65 % | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | *PLUS pH: 8.5 / 手法: 蒸気拡散法, ハンギングドロップ法 | ||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 放射光源 | 波長: 1.5418 |
|---|---|
| 検出器 | タイプ: XUONG-HAMLIN MULTIWIRE / 検出器: AREA DETECTOR / 日付: 1994年8月16日 |
| 放射 | 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.5418 Å / 相対比: 1 |
| 反射 | Num. obs: 19729 / % possible obs: 93.6 % / Observed criterion σ(I): 3 / 冗長度: 2.4 % / Rmerge(I) obs: 0.045 |
| 反射 | *PLUS 最高解像度: 1.75 Å / Rmerge(I) obs: 0.045 |
| 反射 シェル | *PLUS % possible obs: 89.3 % / Rmerge(I) obs: 0.154 / Mean I/σ(I) obs: 5 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 1.75→8 Å / σ(F): 0 詳細: UNUSUAL DIHEDRAL ANGLES (PHI= -140.17 AND PSI= -120.83) ARE OBSERVED FOR SER 164 WHICH MAY ACCOUNT FOR THE CLOSE CONTACT AS AN ACTIVE SITE RESIDUE AND IS PROPOSED TO BE INVOLVED IN THE ...詳細: UNUSUAL DIHEDRAL ANGLES (PHI= -140.17 AND PSI= -120.83) ARE OBSERVED FOR SER 164 WHICH MAY ACCOUNT FOR THE CLOSE CONTACT AS AN ACTIVE SITE RESIDUE AND IS PROPOSED TO BE INVOLVED IN THE CATALYSIS OF METHYL ESTER HYDROLYSIS. PHI/PSI ANGLES THAT LIE OUTSIDE THE EXPECTED RANGE HAVE BEEN OBSERVED FOR MANY OTHER ACTIVE SITE SERINE NUCLEOPHILES IN HYDROLYTIC ENZYMES OF THIS CLASS.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 13.19 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.75→8 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: ARP/PROLSQ/X-PLOR / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj



