+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1cb9 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | NMR STRUCTURE WITH TIGHTLY BOUND WATER MOLECULES OF CYTOTOXIN II (CARDIOTOXIN) FROM NAJA NAJA OXIANA IN AQUEOUS SOLUTION (MAJOR FORM). | ||||||
 要素 要素 | PROTEIN (CYTOTOXIN 2) | ||||||
 キーワード キーワード | TOXIN / CYTOXIN (CARDIOTOXIN) / MEMBRANE PERTURBATION / CIS/TRANS ISOMERIZATION / BOUND WATER | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報other organism cell membrane / toxin activity / killing of cells of another organism / extracellular region / membrane 類似検索 - 分子機能 | ||||||
| 生物種 |  Naja oxiana (コブラ) Naja oxiana (コブラ) | ||||||
| 手法 | 溶液NMR / SIMULATED ANNEALING BY MOLECULAR DYNAMICS IN TORSION ANGLE SPACE | ||||||
 データ登録者 データ登録者 | Dementieva, D.V. / Bocharov, E.V. / Arseniev, A.S. | ||||||
 引用 引用 |  ジャーナル: Eur.J.Biochem. / 年: 1999 ジャーナル: Eur.J.Biochem. / 年: 1999タイトル: Two forms of cytotoxin II (cardiotoxin) from Naja naja oxiana in aqueous solution: spatial structures with tightly bound water molecules. 著者: Dementieva, D.V. / Bocharov, E.V. / Arseniev, A.S. #1:  ジャーナル: RUSS.J.BIOORGANIC CHEM. / 年: 1996 ジャーナル: RUSS.J.BIOORGANIC CHEM. / 年: 1996タイトル: Secondary Structure and Conformational Heterogeneity of Cytotoxin II from Naja Naja Oxiana 著者: Dementieva, D.V. / Utkin Yu, N. / Arseniev, A.S. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1cb9.cif.gz 1cb9.cif.gz | 370.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1cb9.ent.gz pdb1cb9.ent.gz | 310.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1cb9.json.gz 1cb9.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1cb9_validation.pdf.gz 1cb9_validation.pdf.gz | 339.5 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1cb9_full_validation.pdf.gz 1cb9_full_validation.pdf.gz | 444.1 KB | 表示 | |
| XML形式データ |  1cb9_validation.xml.gz 1cb9_validation.xml.gz | 19 KB | 表示 | |
| CIF形式データ |  1cb9_validation.cif.gz 1cb9_validation.cif.gz | 30.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/cb/1cb9 https://data.pdbj.org/pub/pdb/validation_reports/cb/1cb9 ftp://data.pdbj.org/pub/pdb/validation_reports/cb/1cb9 ftp://data.pdbj.org/pub/pdb/validation_reports/cb/1cb9 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 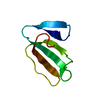
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 6648.238 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Naja oxiana (コブラ) / Secretion: VENOM / 参照: UniProt: P01441 Naja oxiana (コブラ) / Secretion: VENOM / 参照: UniProt: P01441 |
|---|---|
| #2: 水 | ChemComp-HOH / |
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
| ||||||||||||||||||||
| NMR実験の詳細 | Text: THE STRUCTURE WAS DETERMINED BY HOMONUCLEAR NMR SPECTROSCOPY, USING GRADIENT TECHNIQUE. ROESY EXPERIMENTS WERE HELD AT 290 AND 318K. |
- 試料調製
試料調製
| 詳細 | 内容: 90% WATER/10% D2O; 100% D2O |
|---|---|
| 試料状態 | pH: 5.5 / 温度: 303 K |
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
-NMR測定
| NMRスペクトロメーター | タイプ: Varian UNITY 600 / 製造業者: Varian / モデル: UNITY 600 / 磁場強度: 600 MHz |
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: SIMULATED ANNEALING BY MOLECULAR DYNAMICS IN TORSION ANGLE SPACE ソフトェア番号: 1 詳細: THE RESTRAINED REFINMENT WAS MADE IN VACUUM AND ONLY FOR PROTEIN SIDE CHAINS. | ||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: LEAST RESTRAINT VIOLATION (TARGET FUNCTION VALUE) 計算したコンフォーマーの数: 220 / 登録したコンフォーマーの数: 20 |
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj


