+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1c2q | ||||||
|---|---|---|---|---|---|---|---|
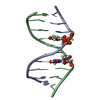
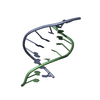
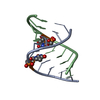
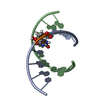
| タイトル | SOLUTION STRUCTURE OF A DNA.RNA HYBRID CONTAINING AN ALPHAT-ANOMERIC THYMIDINE AND POLARITY REVERSALS | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | DNA-RNA HYBRID / DNA/RNA HYBRID / ALPHAT-ANOMERIC THYMIDINE / 3'-3'/5'-5' PHOSPHODIESTER LINKAGES | ||||||
| 機能・相同性 | DNA / RNA 機能・相同性情報 機能・相同性情報 | ||||||
| 手法 | 溶液NMR / SIMULATED ANNEALING, MOLECULAR DYNAMICS | ||||||
| Model type details | minimized average | ||||||
 データ登録者 データ登録者 | Germann, M.W. / Aramini, J.M. | ||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 1999 ジャーナル: Biochemistry / 年: 1999タイトル: Solution structure of a DNA.RNA hybrid containing an alpha-anomeric thymidine and polarity reversals: d(ATGG-3'-3'-alphaT-5'-5'-GCTC). r(gagcaccau). 著者: Aramini, J.M. / Germann, M.W. #1:  ジャーナル: Nucleic Acids Res. / 年: 1998 ジャーナル: Nucleic Acids Res. / 年: 1998タイトル: NMR solution structures of [d(GCGAAT-3'-3'-(alpha-T)-5'-5'-CGC)2] and its unmodified control 著者: Aramini, J.M. / Mujeeb, A. / Germann, M.W. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1c2q.cif.gz 1c2q.cif.gz | 23.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1c2q.ent.gz pdb1c2q.ent.gz | 13.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1c2q.json.gz 1c2q.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1c2q_validation.pdf.gz 1c2q_validation.pdf.gz | 307.9 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1c2q_full_validation.pdf.gz 1c2q_full_validation.pdf.gz | 307.7 KB | 表示 | |
| XML形式データ |  1c2q_validation.xml.gz 1c2q_validation.xml.gz | 2.2 KB | 表示 | |
| CIF形式データ |  1c2q_validation.cif.gz 1c2q_validation.cif.gz | 2.5 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/c2/1c2q https://data.pdbj.org/pub/pdb/validation_reports/c2/1c2q ftp://data.pdbj.org/pub/pdb/validation_reports/c2/1c2q ftp://data.pdbj.org/pub/pdb/validation_reports/c2/1c2q | HTTPS FTP |
-関連構造データ
| 関連構造データ | |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: DNA鎖 | 分子量: 2746.809 Da / 分子数: 1 / 由来タイプ: 合成 / 詳細: SYNTHETIC |
|---|---|
| #2: RNA鎖 | 分子量: 2854.782 Da / 分子数: 1 / 由来タイプ: 合成 / 詳細: SYNTHETIC |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
| ||||||||||||||||||||
| NMR実験の詳細 | Text: THIS STRUCTURE WAS DETERMINED USING STANDARD 2D HOMONUCLEAR NMR TECHNIQUES |
- 試料調製
試料調製
| 詳細 |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料状態 |
| ||||||||||||
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
-NMR測定
| NMRスペクトロメーター | タイプ: Bruker AMX / 製造業者: Bruker / モデル: AMX / 磁場強度: 600 MHz |
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: SIMULATED ANNEALING, MOLECULAR DYNAMICS / ソフトェア番号: 1 詳細: THE STRUCTURE IS BASED ON A TOTAL OF 405 RESTRAINTS: 268 RANDMARDI DERIVED INTERPROTON DISTANCE RESTRAINTS (NON-EXCHANGEABLE: 246; EXCHANGEABLE: 22); 45 DEOXYRIBOSE ENDOCYCLIC TORSION ANGLE ...詳細: THE STRUCTURE IS BASED ON A TOTAL OF 405 RESTRAINTS: 268 RANDMARDI DERIVED INTERPROTON DISTANCE RESTRAINTS (NON-EXCHANGEABLE: 246; EXCHANGEABLE: 22); 45 DEOXYRIBOSE ENDOCYCLIC TORSION ANGLE RESTRAINTS DERIVED FROM PSEUDOROTATION ANALYSIS; 45 RIBOSE ENDOCYCLIC TORSION ANGLE RESTRAINTS (BROAD, N-TYPE); 46 WATSON-CRICK DISTANCE AND ANGLE RESTRAINTS. THE FINAL AVERAGE STRUCTURE WAS OBTAINED BY COORDINATE AVERAGING OF THE FINAL ENSEMBLE OF RMD/REM STRUCTURES, FOLLOWED BY RESTRAINED ENERGY MINIMIZATION. ALL STRUCTURE CALCULATIONS WERE PERFORMED USING THE SANDER PROGRAM WITHIN AMBER 4.1, AND THE 1994 ALL ATOM NUCLEIC ACID PARAMETERIZATION. ALL CALCULATIONS WERE CONDUCTED IN VACUO, USING A DISTANCE DEPENDENT DIELECTRIC CONSTANT AND 30 A CUT-OFF FOR NON-BONDED INTERACTIONS. | ||||||||||||||||||||||||||||||||||||
| 代表構造 | 選択基準: minimized average structure | ||||||||||||||||||||||||||||||||||||
| NMRアンサンブル | 登録したコンフォーマーの数: 1 |
 ムービー
ムービー コントローラー
コントローラー





 PDBj
PDBj Amber
Amber