+ データを開く
データを開く
- 基本情報
基本情報
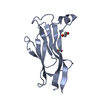
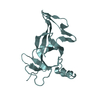
| 登録情報 | データベース: PDB / ID: 1beo | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | BETA-CRYPTOGEIN | ||||||
 要素 要素 | BETA-CRYPTOGEIN | ||||||
 キーワード キーワード | FUNGAL TOXIC ELICITOR / ELICITIN / TOXIN / PLANT PATHOGEN | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont-mediated killing of host cell / symbiont-mediated perturbation of host programmed cell death / extracellular region 類似検索 - 分子機能 | ||||||
| 生物種 |  Phytophthora cryptogea (真核生物) Phytophthora cryptogea (真核生物) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  多波長異常分散, NATIVE DATA / 解像度: 2.2 Å 多波長異常分散, NATIVE DATA / 解像度: 2.2 Å | ||||||
 データ登録者 データ登録者 | Boissy, G. / De La Fortelle, E. / Kahn, R. / Huet, J.C. / Bricogne, G. / Pernollet, J.C. / Brunie, S. | ||||||
 引用 引用 |  ジャーナル: Structure / 年: 1996 ジャーナル: Structure / 年: 1996タイトル: Crystal structure of a fungal elicitor secreted by Phytophthora cryptogea, a member of a novel class of plant necrotic proteins. 著者: Boissy, G. / de La Fortelle, E. / Kahn, R. / Huet, J.C. / Bricogne, G. / Pernollet, J.C. / Brunie, S. #1:  ジャーナル: J.Mol.Biol. / 年: 1993 ジャーナル: J.Mol.Biol. / 年: 1993タイトル: Crystallization and Preliminary X-Ray Diffraction Studies of Beta-Cryptogein, a Toxic Elicitin Secreted by the Phytopathogenic Fungus Phytophthora Cryptogea 著者: Guilloteau, J.P. / Nespoulous, C. / Huet, J.C. / Beauvais, F. / Pernollet, J.C. / Brunie, S. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1beo.cif.gz 1beo.cif.gz | 29.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1beo.ent.gz pdb1beo.ent.gz | 19.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1beo.json.gz 1beo.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/be/1beo https://data.pdbj.org/pub/pdb/validation_reports/be/1beo ftp://data.pdbj.org/pub/pdb/validation_reports/be/1beo ftp://data.pdbj.org/pub/pdb/validation_reports/be/1beo | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 10340.815 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Phytophthora cryptogea (真核生物) / 参照: UniProt: P15570 Phytophthora cryptogea (真核生物) / 参照: UniProt: P15570 |
|---|---|
| #2: 水 | ChemComp-HOH / |
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.7 Å3/Da / 溶媒含有率: 66 % 解説: PHASING: 4 WAVELENGTHS MAD DATA COLLECTION IN THE VICINITY OF THE PLATINUM LIII ABSORPTION EDGE (ESRF D2AM, 26-08-95). | |||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | pH: 7.5 / 詳細: 4.85 M NACL, PH 7.5 | |||||||||||||||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 手法: 蒸気拡散法, ハンギングドロップ法 / 詳細: used to seeding / PH range low: 10 / PH range high: 4 | |||||||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 277 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: LURE シンクロトロン / サイト: LURE  / ビームライン: DW32 / 波長: 0.9 / ビームライン: DW32 / 波長: 0.9 |
| 検出器 | タイプ: MARRESEARCH / 検出器: IMAGE PLATE / 日付: 1995年6月22日 |
| 放射 | 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.9 Å / 相対比: 1 |
| 反射 | Num. obs: 7864 / % possible obs: 97.2 % / Observed criterion σ(I): 3 / 冗長度: 3.1 % / Biso Wilson estimate: 38 Å2 / Rsym value: 0.032 |
| 反射 | *PLUS 最高解像度: 2.2 Å / Num. measured all: 35101 / Rmerge(I) obs: 0.032 |
| 反射 シェル | *PLUS Rmerge(I) obs: 0.15 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  多波長異常分散, NATIVE DATA / 解像度: 2.2→7 Å / σ(F): 2 多波長異常分散, NATIVE DATA / 解像度: 2.2→7 Å / σ(F): 2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 37.3 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.2→7 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称:  X-PLOR / バージョン: 3.1 / 分類: refinement X-PLOR / バージョン: 3.1 / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS Num. reflection all: 7583 / Num. reflection Rfree: 596 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS |
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj
