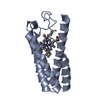
登録情報 データベース : PDB / ID : 1bciタイトル C2 DOMAIN OF CYTOSOLIC PHOSPHOLIPASE A2, NMR, MINIMIZED AVERAGE STRUCTURE CYTOSOLIC PHOSPHOLIPASE A2 キーワード / / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / データ登録者 Xu, G.Y. / Mcdonagh, T. / Yu, H.A. / Nalefski, E.A. / Clark, J.D. / Cumming, D.A. ジャーナル : J.Mol.Biol. / 年 : 1998タイトル : Solution structure and membrane interactions of the C2 domain of cytosolic phospholipase A2.著者 : Xu, G.Y. / McDonagh, T. / Yu, H.A. / Nalefski, E.A. / Clark, J.D. / Cumming, D.A. 履歴 登録 1998年4月30日 処理サイト 改定 1.0 1998年11月25日 Provider / タイプ 改定 1.1 2008年3月24日 Group 改定 1.2 2011年7月13日 Group 改定 1.3 2022年2月16日 Group / Derived calculations / Otherカテゴリ database_2 / pdbx_database_status ... database_2 / pdbx_database_status / pdbx_struct_assembly / pdbx_struct_conn_angle / pdbx_struct_oper_list / struct_conn / struct_site Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.process_site / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id 改定 1.4 2024年5月22日 Group / カテゴリ / chem_comp_bond
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 引用
引用 ジャーナル: J.Mol.Biol. / 年: 1998
ジャーナル: J.Mol.Biol. / 年: 1998 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1bci.cif.gz
1bci.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1bci.ent.gz
pdb1bci.ent.gz PDB形式
PDB形式 1bci.json.gz
1bci.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 1bci_validation.pdf.gz
1bci_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 1bci_full_validation.pdf.gz
1bci_full_validation.pdf.gz 1bci_validation.xml.gz
1bci_validation.xml.gz 1bci_validation.cif.gz
1bci_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/bc/1bci
https://data.pdbj.org/pub/pdb/validation_reports/bc/1bci ftp://data.pdbj.org/pub/pdb/validation_reports/bc/1bci
ftp://data.pdbj.org/pub/pdb/validation_reports/bc/1bci リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 細胞株: PROPRIETARY STRAIN/GI400
Homo sapiens (ヒト) / 細胞株: PROPRIETARY STRAIN/GI400
 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj







 X-PLOR
X-PLOR