+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1b2i | ||||||
|---|---|---|---|---|---|---|---|
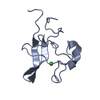
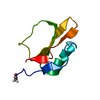
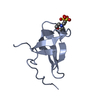
| タイトル | KRINGLE 2 DOMAIN OF HUMAN PLASMINOGEN: NMR SOLUTION STRUCTURE OF TRANS-4-AMINOMETHYLCYCLOHEXANE-1-CARBOXYLIC ACID (AMCHA) COMPLEX | ||||||
 要素 要素 | PROTEIN (PLASMINOGEN) | ||||||
 キーワード キーワード | HYDROLASE / SERINE PROTEASE / FIBRINOLYSIS / LYSINE-BINDING DOMAIN / PLASMINOGEN / KRINGLE 2 | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報plasmin / tissue remodeling / protein antigen binding / Signaling by PDGF / positive regulation of fibrinolysis / negative regulation of cell-cell adhesion mediated by cadherin / Dissolution of Fibrin Clot / biological process involved in interaction with symbiont / Activation of Matrix Metalloproteinases / apolipoprotein binding ...plasmin / tissue remodeling / protein antigen binding / Signaling by PDGF / positive regulation of fibrinolysis / negative regulation of cell-cell adhesion mediated by cadherin / Dissolution of Fibrin Clot / biological process involved in interaction with symbiont / Activation of Matrix Metalloproteinases / apolipoprotein binding / extracellular matrix disassembly / negative regulation of cell-substrate adhesion / positive regulation of blood vessel endothelial cell migration / negative regulation of fibrinolysis / fibrinolysis / Degradation of the extracellular matrix / serine-type peptidase activity / platelet alpha granule lumen / kinase binding / Regulation of Insulin-like Growth Factor (IGF) transport and uptake by Insulin-like Growth Factor Binding Proteins (IGFBPs) / blood coagulation / Platelet degranulation / protein-folding chaperone binding / protease binding / : / endopeptidase activity / blood microparticle / protein domain specific binding / external side of plasma membrane / signaling receptor binding / negative regulation of cell population proliferation / serine-type endopeptidase activity / enzyme binding / cell surface / proteolysis / extracellular space / extracellular exosome / extracellular region / plasma membrane 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 | 溶液NMR / DISTANCE GEOMETRY, DYNAMIC SIMULATED ANNEALING | ||||||
 データ登録者 データ登録者 | Marti, D.N. / Schaller, J. / Llinas, M. | ||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 1999 ジャーナル: Biochemistry / 年: 1999タイトル: Solution structure and dynamics of the plasminogen kringle 2-AMCHA complex: 3(1)-helix in homologous domains. 著者: Marti, D.N. / Schaller, J. / Llinas, M. #1:  ジャーナル: Biochemistry / 年: 1997 ジャーナル: Biochemistry / 年: 1997タイトル: Ligand Preferences of Kringle 2 and Homologous Domains of Human Plasminogen: Canvassing Weak, Intermediate and High-Affinity Binding Sites by 1H-NMR 著者: Marti, D.N. / Hu, C.K. / An, S.S.A. / Von Haller, P. / Schaller, J. / Llinas, M. #2:  ジャーナル: Biochemistry / 年: 1996 ジャーナル: Biochemistry / 年: 1996タイトル: Recombinant Gene Expression and 1H NMR Characteristics of the Kringle (2+3) Supermodule: Spectroscopic/Functional Individuality of Plasminogen Kringle Domains 著者: Soehndel, S. / Hu, C.K. / Marti, D. / Affolter, M. / Schaller, J. / Llinas, M. / Rickli, E.E. #3:  ジャーナル: Eur.J.Biochem. / 年: 1994 ジャーナル: Eur.J.Biochem. / 年: 1994タイトル: Expression, Purification and Characterization of the Recombinant Kringle 2 and Kringle 3 Domains of Human Plasminogen and Analysis of Their Binding Affinity for Omega-Aminocarboxylic Acids 著者: Marti, D. / Schaller, J. / Ochensberger, B. / Rickli, E.E. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1b2i.cif.gz 1b2i.cif.gz | 515.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1b2i.ent.gz pdb1b2i.ent.gz | 430.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1b2i.json.gz 1b2i.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/b2/1b2i https://data.pdbj.org/pub/pdb/validation_reports/b2/1b2i ftp://data.pdbj.org/pub/pdb/validation_reports/b2/1b2i ftp://data.pdbj.org/pub/pdb/validation_reports/b2/1b2i | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 9651.812 Da / 分子数: 1 / 断片: KRINGLE 2 DOMAIN / 変異: C181T, E182S, C188G / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / プラスミド: PQE-8 / 生物種 (発現宿主): Escherichia coli / 発現宿主: Homo sapiens (ヒト) / プラスミド: PQE-8 / 生物種 (発現宿主): Escherichia coli / 発現宿主:  |
|---|---|
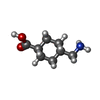
| #2: 化合物 | ChemComp-AMH / |
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 | 内容: 90% H2O/10% D2O |
|---|---|
| 試料状態 | イオン強度: SEE PAPER / pH: 5.1 / 圧: AMBIENT / 温度: 310 K |
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| NMR software |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: DISTANCE GEOMETRY, DYNAMIC SIMULATED ANNEALING / ソフトェア番号: 1 | ||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: TOTAL ENERGY, RAMACHANDRAN PLOT, LEAST RESTRAINT VIOLATION 計算したコンフォーマーの数: 150 / 登録したコンフォーマーの数: 20 |
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj





 HSQC
HSQC