+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1axp | ||||||
|---|---|---|---|---|---|---|---|
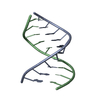
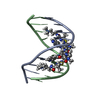
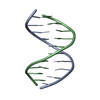
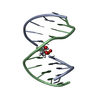
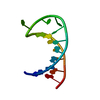
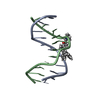
| タイトル | DNA DUPLEX CONTAINING A PURINE-RICH STRAND, NMR, 6 STRUCTURES | ||||||
 要素 要素 | (DNA (D(GAAGAGAAGC)(DOT)D(GCTTCTCTTC))) x 2 | ||||||
 キーワード キーワード | DNA / DNA DUPLEX / PURINE/PYRIMIDINE-RICH STRANDS / B-FORM / DEOXYRIBONUCLEIC ACID | ||||||
| 機能・相同性 | DNA 機能・相同性情報 機能・相同性情報 | ||||||
| 手法 | 溶液NMR / simulated annealing | ||||||
 データ登録者 データ登録者 | Gyi, J.I. / Lane, A.N. / Conn, G.L. / Brown, T. | ||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 1998 ジャーナル: Biochemistry / 年: 1998タイトル: Solution structures of DNA.RNA hybrids with purine-rich and pyrimidine-rich strands: comparison with the homologous DNA and RNA duplexes. 著者: Gyi, J.I. / Lane, A.N. / Conn, G.L. / Brown, T. #1:  ジャーナル: Biochemistry / 年: 1996 ジャーナル: Biochemistry / 年: 1996タイトル: Comparison of the Thermodynamic Stabilities and Solution Conformations of DNA.RNA Hybrids Containing Purine-Rich and Pyrimidine-Rich Strands with DNA and RNA Duplexes 著者: Gyi, J.I. / Conn, G.L. / Lane, A.N. / Brown, T. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1axp.cif.gz 1axp.cif.gz | 80.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1axp.ent.gz pdb1axp.ent.gz | 64.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1axp.json.gz 1axp.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1axp_validation.pdf.gz 1axp_validation.pdf.gz | 311.1 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1axp_full_validation.pdf.gz 1axp_full_validation.pdf.gz | 348.8 KB | 表示 | |
| XML形式データ |  1axp_validation.xml.gz 1axp_validation.xml.gz | 2.2 KB | 表示 | |
| CIF形式データ |  1axp_validation.cif.gz 1axp_validation.cif.gz | 3.4 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ax/1axp https://data.pdbj.org/pub/pdb/validation_reports/ax/1axp ftp://data.pdbj.org/pub/pdb/validation_reports/ax/1axp ftp://data.pdbj.org/pub/pdb/validation_reports/ax/1axp | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: DNA鎖 | 分子量: 3127.082 Da / 分子数: 1 / 由来タイプ: 合成 |
|---|---|
| #2: DNA鎖 | 分子量: 2961.940 Da / 分子数: 1 / 由来タイプ: 合成 |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
| ||||||||||||
| NMR実験の詳細 | Text: THE ASSIGNMENTS, NOES AND COUPLING CONSTANTS DETERMINED FROM 2D HOMONUCLEAR NMR EXPERIMENTS USING UNLABELLED SAMPLE. |
- 試料調製
試料調製
| 試料状態 | pH: 7 / 温度: 303 K |
|---|---|
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| ソフトウェア | 名称:  AMBER / 分類: 精密化 AMBER / 分類: 精密化 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR software |
| ||||||||||||||||||||||||
| 精密化 | 手法: simulated annealing / ソフトェア番号: 1 詳細: DISTANCE CONSTRAINTS FOR GLYCOSIDIC TORSION ANGLES, DEOXYRIBOSE PSEUDOROTATIONAL PHASE ANGLES AND AMPLITUDES CALCULATED FROM NOE BUILD-UP CURVES AND COUPLING CONSTANTS BY A LEAST SQUARES/GRID- ...詳細: DISTANCE CONSTRAINTS FOR GLYCOSIDIC TORSION ANGLES, DEOXYRIBOSE PSEUDOROTATIONAL PHASE ANGLES AND AMPLITUDES CALCULATED FROM NOE BUILD-UP CURVES AND COUPLING CONSTANTS BY A LEAST SQUARES/GRID-SEARCH METHOD IMPLEMENTED IN NUCFIT AND PFIT (A.N. LANE, NIMR, UK). INTERNUCLEOTIDE DISTANCE CONSTRAINTS CALCULATED FROM NOE BUILD-UP CURVES. STRUCTURES CALCULATED BY SIMULATED ANNEALING/RMD PROTOCOL WITHIN DISCOVER 95.0 USING AN AMBER FORCEFIELD AND A DISTANCE DEPENDENT DIELECTRIC CONSTANT. | ||||||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: LOWEST POTENTIAL ENERGY AND MINIMAL VIOLATIONS AND ACCEPTABLE STEREOCHEMISTRY 計算したコンフォーマーの数: 32 / 登録したコンフォーマーの数: 6 |
 ムービー
ムービー コントローラー
コントローラー







 PDBj
PDBj

