+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1aju | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | HIV-2 TAR-ARGININAMIDE COMPLEX, NMR, 20 STRUCTURES | ||||||
 要素 要素 | TAR RNA | ||||||
 キーワード キーワード | RNA / COMPLEX (RIBONUCLEIC ACID-LIGAND) / TRANSCRIPTIONAL ACTIVATION / PROTEIN-RNA INTERACTIONS | ||||||
| 機能・相同性 | ARGININE / RNA / RNA (> 10) 機能・相同性情報 機能・相同性情報 | ||||||
| 手法 | 溶液NMR / RANDOM COORDINATES, SIMULATED ANNEALING | ||||||
 データ登録者 データ登録者 | Brodsky, A.S. / Williamson, J.R. | ||||||
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 1997 ジャーナル: J.Mol.Biol. / 年: 1997タイトル: Solution structure of the HIV-2 TAR-argininamide complex. 著者: Brodsky, A.S. / Williamson, J.R. #1:  ジャーナル: J.Am.Chem.Soc. / 年: 1996 ジャーナル: J.Am.Chem.Soc. / 年: 1996タイトル: Geometric Parameters in Nucleic Acids: Sugar and Phosphate Constituents 著者: Gelbin, A. / Schneider, B. / Clowny, L. / Hsieh, S.-H. / Olsen, W.K. / Berman, H.M. #2:  ジャーナル: J.Mol.Biol. / 年: 1995 ジャーナル: J.Mol.Biol. / 年: 1995タイトル: The Structure of the Human Immunodeficiency Virus Type-1 Tar RNA Reveals Principles of RNA Recognition by Tat Protein 著者: Aboul-Ela, F. / Karn, J. / Varani, G. #3:  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1993 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1993タイトル: Role of RNA Structure in Arginine Recognition of Tar RNA 著者: Puglisi, J.D. / Chen, L. / Frankel, A.D. / Williamson, J.R. #4:  ジャーナル: Science / 年: 1992 ジャーナル: Science / 年: 1992タイトル: Conformation of the Tar RNA-Arginine Complex by NMR Spectroscopy 著者: Puglisi, J.D. / Tan, R. / Calnan, B.J. / Frankel, A.D. / Williamson, J.R. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1aju.cif.gz 1aju.cif.gz | 380.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1aju.ent.gz pdb1aju.ent.gz | 320.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1aju.json.gz 1aju.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1aju_validation.pdf.gz 1aju_validation.pdf.gz | 334.7 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1aju_full_validation.pdf.gz 1aju_full_validation.pdf.gz | 450.2 KB | 表示 | |
| XML形式データ |  1aju_validation.xml.gz 1aju_validation.xml.gz | 8.8 KB | 表示 | |
| CIF形式データ |  1aju_validation.cif.gz 1aju_validation.cif.gz | 15.6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/aj/1aju https://data.pdbj.org/pub/pdb/validation_reports/aj/1aju ftp://data.pdbj.org/pub/pdb/validation_reports/aj/1aju ftp://data.pdbj.org/pub/pdb/validation_reports/aj/1aju | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 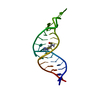
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: RNA鎖 | 分子量: 9652.761 Da / 分子数: 1 / 由来タイプ: 合成 |
|---|---|
| #2: 化合物 | ChemComp-ARG / |
-実験情報
-実験
| 実験 | 手法: 溶液NMR |
|---|---|
| NMR実験 | タイプ: SEE PAPER |
- 試料調製
試料調製
| 試料状態 | pH: 6.4 / 温度: 298 K |
|---|---|
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| ソフトウェア |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR software |
| ||||||||||||
| 精密化 | 手法: RANDOM COORDINATES, SIMULATED ANNEALING / ソフトェア番号: 1 詳細: BOND LENGTHS AND ANGLES WERE USED AS DESCRIBED BY THE NUCLEIC ACID DATABASE (SEE REFERENCE REFINEMENT DETAILS CAN BE FOUND IN THE JRNL CITATION ABOVE. | ||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: LEAST RESTRAINTS AND LOWEST ENERGY 計算したコンフォーマーの数: 20 / 登録したコンフォーマーの数: 20 |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj
































 X-PLOR
X-PLOR