+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9953 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structural insights into stressosome assembly | |||||||||
 マップデータ マップデータ | RsbRA/S complex | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
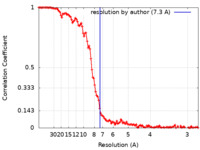
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.3 Å | |||||||||
 データ登録者 データ登録者 | Kim H / Kwon E / Pathak D / Kim DY / Jung HS | |||||||||
| 資金援助 |  韓国, 2件 韓国, 2件
| |||||||||
 引用 引用 | ジャーナル: J Mol Biol / 年: 2011 タイトル: Role of the tail in the regulated state of myosin 2. 著者: Hyun Suk Jung / Neil Billington / Kavitha Thirumurugan / Bridget Salzameda / Christine R Cremo / Joseph M Chalovich / Peter D Chantler / Peter J Knight /  要旨: Myosin 2 from vertebrate smooth muscle or non-muscle sources is in equilibrium between compact, inactive monomers and thick filaments under physiological conditions. In the inactive monomer, the two ...Myosin 2 from vertebrate smooth muscle or non-muscle sources is in equilibrium between compact, inactive monomers and thick filaments under physiological conditions. In the inactive monomer, the two heads pack compactly together, and the long tail is folded into three closely packed segments that are associated chiefly with one of the heads. The molecular basis of the folding of the tail remains unexplained. By using electron microscopy, we show that compact monomers of smooth muscle myosin 2 have the same structure in both the native state and following specific, intramolecular photo-cross-linking between Cys109 of the regulatory light chain (RLC) and segment 3 of the tail. Nonspecific cross-linking between lysine residues of the folded monomer by glutaraldehyde also does not perturb the compact conformation and stabilizes it against unfolding at high ionic strength. Sequence comparisons across phyla and myosin 2 isoforms suggest that the folding of the tail is stabilized by ionic interactions between the positively charged N-terminal sequence of the RLC and a negatively charged region near the start of tail segment 3 and that phosphorylation of the RLC could perturb these interactions. Our results support the view that interactions between the heads and the distal tail perform a critical role in regulating activity of myosin 2 molecules through stabilizing the compact monomer conformation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9953.map.gz emd_9953.map.gz | 10.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9953-v30.xml emd-9953-v30.xml emd-9953.xml emd-9953.xml | 18.7 KB 18.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_9953_fsc.xml emd_9953_fsc.xml | 13.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9953.png emd_9953.png | 206.8 KB | ||
| マスクデータ |  emd_9953_msk_1.map emd_9953_msk_1.map | 137.1 MB |  マスクマップ マスクマップ | |
| その他 |  emd_9953_half_map_1.map.gz emd_9953_half_map_1.map.gz emd_9953_half_map_2.map.gz emd_9953_half_map_2.map.gz | 105.8 MB 105.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9953 http://ftp.pdbj.org/pub/emdb/structures/EMD-9953 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9953 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9953 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9953_validation.pdf.gz emd_9953_validation.pdf.gz | 447.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9953_full_validation.pdf.gz emd_9953_full_validation.pdf.gz | 447.4 KB | 表示 | |
| XML形式データ |  emd_9953_validation.xml.gz emd_9953_validation.xml.gz | 19.1 KB | 表示 | |
| CIF形式データ |  emd_9953_validation.cif.gz emd_9953_validation.cif.gz | 25 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9953 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9953 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9953 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9953 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9953.map.gz / 形式: CCP4 / 大きさ: 137.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9953.map.gz / 形式: CCP4 / 大きさ: 137.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RsbRA/S complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.4 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
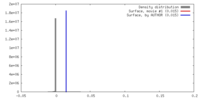
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_9953_msk_1.map emd_9953_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
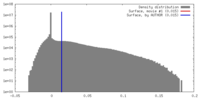
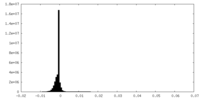
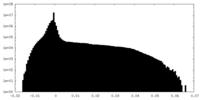
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_9953_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
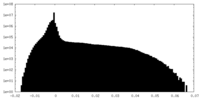
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_9953_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
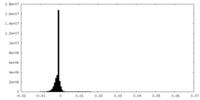
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : RsbRA/RsbS complex from Bacillus subtilis
| 全体 | 名称: RsbRA/RsbS complex from Bacillus subtilis |
|---|---|
| 要素 |
|
-超分子 #1: RsbRA/RsbS complex from Bacillus subtilis
| 超分子 | 名称: RsbRA/RsbS complex from Bacillus subtilis / タイプ: complex / ID: 1 / 親要素: 0 詳細: Stressosome (RsbRA/RsbS) complex from Bacillus subtilis |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Enterobacteria phage L1 (ファージ) / 組換株: BL-21star (DE3) Enterobacteria phage L1 (ファージ) / 組換株: BL-21star (DE3) |
| 分子量 | 実験値: 1.8 MDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: 20 mM HEPES, 50 mM NaCl, 0.2 mM TCEP, 5 % glycerol, pH 7.5 | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 39.2 K / 装置: FEI VITROBOT MARK IV / 詳細: blot for 7 seconds before plunging. | |||||||||||||||
| 詳細 | RsbRA/RsbS complex buffer: 20 mM HEPES, 50 mM NaCl, 0.2 mM TCEP, 5 % glycerol, pH 7.5 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: OTHER / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 実像数: 1788 / 平均露光時間: 1.0 sec. / 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 3.0 µm / 最小 デフォーカス(補正後): 1.5 µm / 倍率(補正後): 47000 / 照射モード: OTHER / 撮影モード: OTHER / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 47000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)