[日本語] English
 万見
万見- EMDB-9350: SegA-asym, conformation of TDP-43 low complexity domain segment A asym -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9350 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | SegA-asym, conformation of TDP-43 low complexity domain segment A asym | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Amyloid / TDP43 / ALS / FTLD-TDP / dna binding protein / protein fibril | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報nuclear inner membrane organization / interchromatin granule / perichromatin fibrils / 3'-UTR-mediated mRNA destabilization / 3'-UTR-mediated mRNA stabilization / intracellular membraneless organelle / negative regulation of protein phosphorylation / host-mediated suppression of viral transcription / pre-mRNA intronic binding / RNA splicing ...nuclear inner membrane organization / interchromatin granule / perichromatin fibrils / 3'-UTR-mediated mRNA destabilization / 3'-UTR-mediated mRNA stabilization / intracellular membraneless organelle / negative regulation of protein phosphorylation / host-mediated suppression of viral transcription / pre-mRNA intronic binding / RNA splicing / response to endoplasmic reticulum stress / mRNA 3'-UTR binding / molecular condensate scaffold activity / regulation of circadian rhythm / positive regulation of insulin secretion / regulation of protein stability / positive regulation of protein import into nucleus / mRNA processing / cytoplasmic stress granule / rhythmic process / regulation of gene expression / double-stranded DNA binding / regulation of apoptotic process / amyloid fibril formation / regulation of cell cycle / nuclear speck / RNA polymerase II cis-regulatory region sequence-specific DNA binding / negative regulation of gene expression / lipid binding / chromatin / mitochondrion / DNA binding / RNA binding / nucleoplasm / identical protein binding / nucleus 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
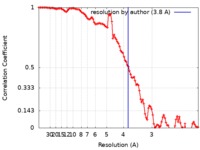
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Cao Q / Boyer DR | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2019 ジャーナル: Nat Struct Mol Biol / 年: 2019タイトル: Cryo-EM structures of four polymorphic TDP-43 amyloid cores. 著者: Qin Cao / David R Boyer / Michael R Sawaya / Peng Ge / David S Eisenberg /  要旨: The DNA and RNA processing protein TDP-43 undergoes both functional and pathogenic aggregation. Functional TDP-43 aggregates form reversible, transient species such as nuclear bodies, stress ...The DNA and RNA processing protein TDP-43 undergoes both functional and pathogenic aggregation. Functional TDP-43 aggregates form reversible, transient species such as nuclear bodies, stress granules, and myo-granules. Pathogenic, irreversible TDP-43 aggregates form in amyotrophic lateral sclerosis and other neurodegenerative conditions. Here we find the features of TDP-43 fibrils that confer both reversibility and irreversibility by determining structures of two segments reported to be the pathogenic cores of human TDP-43 aggregation: SegA (residues 311-360), which forms three polymorphs, all with dagger-shaped folds; and SegB A315E (residues 286-331 containing the amyotrophic lateral sclerosis hereditary mutation A315E), which forms R-shaped folds. Energetic analysis suggests that the dagger-shaped polymorphs represent irreversible fibril structures, whereas the SegB polymorph may participate in both reversible and irreversible fibrils. Our structures reveal the polymorphic nature of TDP-43 and suggest how the A315E mutation converts the R-shaped polymorph to an irreversible form that enhances pathology. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9350.map.gz emd_9350.map.gz | 7.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9350-v30.xml emd-9350-v30.xml emd-9350.xml emd-9350.xml | 10.2 KB 10.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_9350_fsc.xml emd_9350_fsc.xml | 11.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9350.png emd_9350.png | 165.9 KB | ||
| Filedesc metadata |  emd-9350.cif.gz emd-9350.cif.gz | 5.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9350 http://ftp.pdbj.org/pub/emdb/structures/EMD-9350 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9350 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9350 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9350_validation.pdf.gz emd_9350_validation.pdf.gz | 611.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9350_full_validation.pdf.gz emd_9350_full_validation.pdf.gz | 611.4 KB | 表示 | |
| XML形式データ |  emd_9350_validation.xml.gz emd_9350_validation.xml.gz | 11 KB | 表示 | |
| CIF形式データ |  emd_9350_validation.cif.gz emd_9350_validation.cif.gz | 14.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9350 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9350 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9350 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9350 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6n3bMC  0334C  9339C  9349C  6n37C  6n3aC  6n3cC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10497 (タイトル: Cryo-EM structures of four polymorphic TDP-43 amyloid cores EMPIAR-10497 (タイトル: Cryo-EM structures of four polymorphic TDP-43 amyloid coresData size: 4.5 TB Data #1: Unaligned dose fractionated frames of TDP-43 SegA amyloid fibrils [micrographs - multiframe] Data #2: Unaligned dose fractionated frames of TDP-43 SegB A315E amyloid fibrils [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9350.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9350.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.064 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : TDP43 fibril
| 全体 | 名称: TDP43 fibril |
|---|---|
| 要素 |
|
-超分子 #1: TDP43 fibril
| 超分子 | 名称: TDP43 fibril / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: TAR DNA-binding protein 43
| 分子 | 名称: TAR DNA-binding protein 43 / タイプ: protein_or_peptide / ID: 1 / コピー数: 10 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 5.207797 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MNFGAFSINP AMMAAAQAAL QSSWGMMGML ASQQNQSGPS GNNQNQGNMQ UniProtKB: TAR DNA-binding protein 43 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均露光時間: 8.0 sec. / 平均電子線量: 48.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 0.002 µm / 最小 デフォーカス(公称値): 0.002 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-6n3b: |
 ムービー
ムービー コントローラー
コントローラー











 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)