+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9262 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single-Molecule 3D Image of Double Complexes of DNA-Nanogold Conjugates (No. 01) | |||||||||
 マップデータ マップデータ | Double Complexes of DNA-Nanogold Conjugates (No. 01) | |||||||||
 試料 試料 |
| |||||||||
| 生物種 | synthetic construct (人工物) | |||||||||
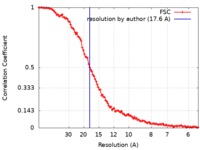
| 手法 | 電子線トモグラフィー法 / ネガティブ染色法 / 解像度: 17.6 Å | |||||||||
 データ登録者 データ登録者 | Wu H / Zhai X / Lei D / Liu J / Yu Y / Bie R / Ren G | |||||||||
 引用 引用 |  ジャーナル: Sci Rep / 年: 2018 ジャーナル: Sci Rep / 年: 2018タイトル: An Algorithm for Enhancing the Image Contrast of Electron Tomography. 著者: Hao Wu / Xiaobo Zhai / Dongsheng Lei / Jianfang Liu / Yadong Yu / Rongfang Bie / Gang Ren /   要旨: Three-dimensional (3D) reconstruction of a single protein molecule is essential for understanding the relationship between the structural dynamics and functions of the protein. Electron tomography ...Three-dimensional (3D) reconstruction of a single protein molecule is essential for understanding the relationship between the structural dynamics and functions of the protein. Electron tomography (ET) provides a tool for imaging an individual particle of protein from a series of tilted angles. Individual-particle electron tomography (IPET) provides an approach for reconstructing a 3D density map from a single targeted protein particle (without averaging from different particles of this type of protein), in which the target particle was imaged from a series of tilting angles. However, owing to radiation damage limitations, low-dose images (high noise, and low image contrast) are often challenging to be aligned for 3D reconstruction at intermediate resolution (1-3 nm). Here, we propose a computational method to enhance the image contrast, without increasing any experimental dose, for IPET 3D reconstruction. Using an edge-preserving smoothing-based multi-scale image decomposition algorithm, this method can detect the object against a high-noise background and enhance the object image contrast without increasing the noise level or significantly decreasing the image resolution. The method was validated by using both negative staining (NS) ET and cryo-ET images. The successful 3D reconstruction of a small molecule (<100 kDa) indicated that this method can be used as a supporting tool to current ET 3D reconstruction methods for studying protein dynamics via structure determination from each individual particle of the same type of protein. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9262.map.gz emd_9262.map.gz | 93.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9262-v30.xml emd-9262-v30.xml emd-9262.xml emd-9262.xml | 11 KB 11 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_9262_fsc.xml emd_9262_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9262.png emd_9262.png | 37.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9262 http://ftp.pdbj.org/pub/emdb/structures/EMD-9262 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9262 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9262 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9262_validation.pdf.gz emd_9262_validation.pdf.gz | 79.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9262_full_validation.pdf.gz emd_9262_full_validation.pdf.gz | 78.4 KB | 表示 | |
| XML形式データ |  emd_9262_validation.xml.gz emd_9262_validation.xml.gz | 495 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9262 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9262 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9262 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9262 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9262.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9262.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Double Complexes of DNA-Nanogold Conjugates (No. 01) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.82 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Double complexes of 84-base pair double-stranded DNA bound with t...
| 全体 | 名称: Double complexes of 84-base pair double-stranded DNA bound with two 5-nm nanogolds |
|---|---|
| 要素 |
|
-超分子 #1: Double complexes of 84-base pair double-stranded DNA bound with t...
| 超分子 | 名称: Double complexes of 84-base pair double-stranded DNA bound with two 5-nm nanogolds タイプ: complex / ID: 1 / 親要素: 0 詳細: 5-nm nanogold particles were stabilized via exchanging with bis-(p-sulfonatophenyl) phenylphosphine (BSPP). DNA sequences modified with a 5 thiol moiety were PAGE purified. DNA thiolated at ...詳細: 5-nm nanogold particles were stabilized via exchanging with bis-(p-sulfonatophenyl) phenylphosphine (BSPP). DNA sequences modified with a 5 thiol moiety were PAGE purified. DNA thiolated at the 5 end was re-suspended in buffer (10mM Tris pH 8, 0.5mM EDTA). Nanogold particles and DNA were combined at a stoichiometric ratio of 1:2 in the presence of a reducing agent. Monoconjugates formed were separated by anion exchange HPLC, and the fractions concentrated by an Amicon Ultra spin filter, MW 100,000 (EMD Millipore Corp, Billerica, MA). Twenty microliters of nanogold monoconjugates, each containing complementary strands of DNA, were combined stoichiometrically as determined by absorption at 520 nm and allowed to react overnight at room temperature. The dimers were purified from unreacted monoconjugates by agarose gel electrophoresis. |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 電子線トモグラフィー法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.02 mg/mL |
|---|---|
| 緩衝液 | pH: 7 詳細: 1X Dulbeccos phosphate-buffered saline, 2.7 mM KCl, 1.46 mM KH2PO4, 136.9 mM NaCl, and 8.1 mM Na2HPO4 |
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Formate 詳細: The grid was stained with 1% (w/v) uranyl formate by using optimized negative-staining (OpNS) protocol. |
| グリッド | モデル: Homemade / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS |
| 詳細 | 5-nm nanogold particles were stabilized via exchanging with bis-(p-sulfonatophenyl) phenylphosphine (BSPP). DNA sequences modified with a 5 thiol moiety were PAGE purified. DNA thiolated at the 5 end was re-suspended in buffer (10mM Tris pH 8, 0.5mM EDTA). Nanogold particles and DNA were combined at a stoichiometric ratio of 1:2 in the presence of a reducing agent. Monoconjugates formed were separated by anion exchange HPLC, and the fractions concentrated by an Amicon Ultra spin filter, MW 100,000 (EMD Millipore Corp, Billerica, MA). Twenty microliters of nanogold monoconjugates, each containing complementary strands of DNA, were combined stoichiometrically as determined by absorption at 520 nm and allowed to react overnight at room temperature. The dimers were purified from unreacted monoconjugates by agarose gel electrophoresis. |
| 切片作成 | その他: NO SECTIONING |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | ZEISS LIBRA120PLUS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 平均電子線量: 54.64 e/Å2 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.2 mm / 倍率(公称値): 125000 |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)