+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9191 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of double-stranded target DNA engaged Csy complex from Pseudomonas aeruginosa (PA-14) | |||||||||
 マップデータ マップデータ | Composite sharpened map of the complex generated from multiple focused maps of different sub-regions of the complex. A B-factor of -35 was used for sharpening the map. | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報maintenance of CRISPR repeat elements / endonuclease activity / defense response to virus / 加水分解酵素; エステル加水分解酵素 / RNA binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌) / Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌) /  Pseudomonas aeruginosa UCBPP-PA14 (緑膿菌) Pseudomonas aeruginosa UCBPP-PA14 (緑膿菌) | |||||||||
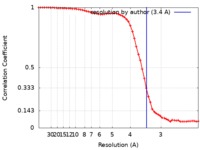
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Chowdhury S / Rollins MF / Carter J / Golden SM / Miettinen HM / Santiago-Frangos A / Faith D / Lawrence MC / Wiedenheft B / Lander GC | |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2019 ジャーナル: Mol Cell / 年: 2019タイトル: Structure Reveals a Mechanism of CRISPR-RNA-Guided Nuclease Recruitment and Anti-CRISPR Viral Mimicry. 著者: MaryClare F Rollins / Saikat Chowdhury / Joshua Carter / Sarah M Golden / Heini M Miettinen / Andrew Santiago-Frangos / Dominick Faith / C Martin Lawrence / Gabriel C Lander / Blake Wiedenheft /  要旨: Bacteria and archaea have evolved sophisticated adaptive immune systems that rely on CRISPR RNA (crRNA)-guided detection and nuclease-mediated elimination of invading nucleic acids. Here, we present ...Bacteria and archaea have evolved sophisticated adaptive immune systems that rely on CRISPR RNA (crRNA)-guided detection and nuclease-mediated elimination of invading nucleic acids. Here, we present the cryo-electron microscopy (cryo-EM) structure of the type I-F crRNA-guided surveillance complex (Csy complex) from Pseudomonas aeruginosa bound to a double-stranded DNA target. Comparison of this structure to previously determined structures of this complex reveals a ∼180-degree rotation of the C-terminal helical bundle on the "large" Cas8f subunit. We show that the double-stranded DNA (dsDNA)-induced conformational change in Cas8f exposes a Cas2/3 "nuclease recruitment helix" that is structurally homologous to a virally encoded anti-CRISPR protein (AcrIF3). Structural homology between Cas8f and AcrIF3 suggests that AcrIF3 is a mimic of the Cas8f nuclease recruitment helix. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9191.map.gz emd_9191.map.gz | 11.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9191-v30.xml emd-9191-v30.xml emd-9191.xml emd-9191.xml | 40.5 KB 40.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_9191_fsc.xml emd_9191_fsc.xml | 6.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9191.png emd_9191.png | 162.4 KB | ||
| マスクデータ |  emd_9191_msk_1.map emd_9191_msk_1.map | 15.6 MB |  マスクマップ マスクマップ | |
| その他 |  emd_9191_additional_1.map.gz emd_9191_additional_1.map.gz emd_9191_additional_2.map.gz emd_9191_additional_2.map.gz emd_9191_additional_3.map.gz emd_9191_additional_3.map.gz emd_9191_additional_4.map.gz emd_9191_additional_4.map.gz emd_9191_additional_5.map.gz emd_9191_additional_5.map.gz emd_9191_additional_6.map.gz emd_9191_additional_6.map.gz emd_9191_half_map_1.map.gz emd_9191_half_map_1.map.gz emd_9191_half_map_2.map.gz emd_9191_half_map_2.map.gz | 13.2 MB 14.5 MB 14.2 MB 13.5 MB 13.3 MB 13.7 MB 13.6 MB 13.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9191 http://ftp.pdbj.org/pub/emdb/structures/EMD-9191 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9191 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9191 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9191_validation.pdf.gz emd_9191_validation.pdf.gz | 724.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9191_full_validation.pdf.gz emd_9191_full_validation.pdf.gz | 724.1 KB | 表示 | |
| XML形式データ |  emd_9191_validation.xml.gz emd_9191_validation.xml.gz | 11.2 KB | 表示 | |
| CIF形式データ |  emd_9191_validation.cif.gz emd_9191_validation.cif.gz | 15.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9191 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9191 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9191 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9191 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9191.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9191.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Composite sharpened map of the complex generated from multiple focused maps of different sub-regions of the complex. A B-factor of -35 was used for sharpening the map. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.15 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
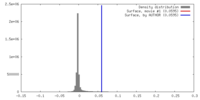
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_9191_msk_1.map emd_9191_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
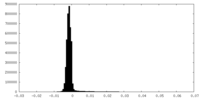
| 密度ヒストグラム |
-追加マップ: Composite unsharpened map of the complex generated from...
| ファイル | emd_9191_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Composite unsharpened map of the complex generated from multiple focused maps of different sub-regions of the complex | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
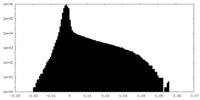
| 密度ヒストグラム |
-追加マップ: Sharpened non-focused map of the full complex
| ファイル | emd_9191_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened non-focused map of the full complex | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
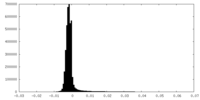
| 密度ヒストグラム |
-追加マップ: Unsharpened non-focused map of the full complex
| ファイル | emd_9191_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened non-focused map of the full complex | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
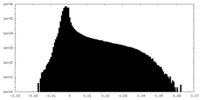
| 密度ヒストグラム |
-追加マップ: Focused map of "Region-1" of the Csy-DNA complex,...
| ファイル | emd_9191_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focused map of "Region-1" of the Csy-DNA complex, comprising the head (Cas6f-crRNA stem loop) - Cas8f C-terminal helix bundle - Cas7.1f and Cas7.2f subunits | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Focused map of "Region-2" of the Csy-DNA complex,...
| ファイル | emd_9191_additional_5.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focused map of "Region-2" of the Csy-DNA complex, comprising all six Cas7f subunits - crRNA - complimentary target DNA strand | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Focused map of "Region-3" of the Csy-DNA complex,...
| ファイル | emd_9191_additional_6.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focused map of "Region-3" of the Csy-DNA complex, comprising the tail (Cas8f N-terminal half - Cas5f - double-stranded target DNA) - Cas7.6 subunit | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Unfiltered composite half map #1
| ファイル | emd_9191_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered composite half map #1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Unfiltered composite half map #2
| ファイル | emd_9191_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered composite half map #2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Double-stranded target DNA engaged Csy Complex from Pseudomonas a...
| 全体 | 名称: Double-stranded target DNA engaged Csy Complex from Pseudomonas aeruginosa (PA-14) |
|---|---|
| 要素 |
|
-超分子 #1: Double-stranded target DNA engaged Csy Complex from Pseudomonas a...
| 超分子 | 名称: Double-stranded target DNA engaged Csy Complex from Pseudomonas aeruginosa (PA-14) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#7 |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌) Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌) |
| 組換発現 | 生物種:  |
| 分子量 | 理論値: 360 KDa |
-分子 #1: CRISPR-associated protein Csy2
| 分子 | 名称: CRISPR-associated protein Csy2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌) Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌)株: UCBPP-PA14 |
| 分子量 | 理論値: 36.244074 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSVTDPEALL LLPRLSIQNA NAISSPLTWG FPSPGAFTGF VHALQRRVGI SLDIELDGVG IVCHRFEAQI SQPAGKRTKV FNLTRNPLN RDGSTAAIVE EGRAHLEVSL LLGVHGDGLD DHPAQEIARQ VQEQAGAMRL AGGSILPWCN ERFPAPNAEL L MLGGSDEQ ...文字列: MSVTDPEALL LLPRLSIQNA NAISSPLTWG FPSPGAFTGF VHALQRRVGI SLDIELDGVG IVCHRFEAQI SQPAGKRTKV FNLTRNPLN RDGSTAAIVE EGRAHLEVSL LLGVHGDGLD DHPAQEIARQ VQEQAGAMRL AGGSILPWCN ERFPAPNAEL L MLGGSDEQ RRKNQRRLTR RLLPGFALVS REALLQQHLE TLRTTLPEAT TLDALLDLCR INFEPPATSS EEEASPPDAA WQ VRDKPGW LVPIPAGYNA LSPLYLPGEV RNARDRETPL RFVENLFGLG EWLSPHRVAA LSDLLWYHHA EPDKGLYRWS TPR FVEHAI A |
-分子 #2: CRISPR-associated protein Csy3
| 分子 | 名称: CRISPR-associated protein Csy3 / タイプ: protein_or_peptide / ID: 2 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌) Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌)株: UCBPP-PA14 |
| 分子量 | 理論値: 37.579273 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSKPILSTAS VLAFERKLDP SDALMSAGAW AQRDASQEWP AVTVREKSVR GTISNRLKTK DRDPAKLDAS IQSPNLQTVD VANLPSDAD TLKVRFTLRV LGGAGTPSAC NDAAYRDKLL QTVATYVNDQ GFAELARRYA HNLANARFLW RNRVGAEAVE V RINHIRQG ...文字列: MSKPILSTAS VLAFERKLDP SDALMSAGAW AQRDASQEWP AVTVREKSVR GTISNRLKTK DRDPAKLDAS IQSPNLQTVD VANLPSDAD TLKVRFTLRV LGGAGTPSAC NDAAYRDKLL QTVATYVNDQ GFAELARRYA HNLANARFLW RNRVGAEAVE V RINHIRQG EVARAWRFDA LAIGLRDFKA DAELDALAEL IASGLSGSGH VLLEVVAFAR IGDGQEVFPS QELILDKGDK KG QKSKTLY SVRDAAAIHS QKIGNALRTI DTWYPDEDGL GPIAVEPYGS VTSQGKAYRQ PKQKLDFYTL LDNWVLRDEA PAV EQQHYV IANLIRGGVF GEAEEK |
-分子 #3: CRISPR-associated endonuclease Cas6/Csy4
| 分子 | 名称: CRISPR-associated endonuclease Cas6/Csy4 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; エステル加水分解酵素 |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌) Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌)株: UCBPP-PA14 |
| 分子量 | 理論値: 21.675781 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: FTMDHYLDIR LRPDPEFPPA QLMSVLFGKL HQALVAQGGD RIGVSFPDLD ESRSRLGERL RIHASADDLR ALLARPWLEG LRDHLQFGE PAVVPHPTPY RQVSRVQAKS NPERLRRRLM RRHDLSEEEA RKRIPDTVAR ALDLPFVTLR SQSTGQHFRL F IRHGPLQV ...文字列: FTMDHYLDIR LRPDPEFPPA QLMSVLFGKL HQALVAQGGD RIGVSFPDLD ESRSRLGERL RIHASADDLR ALLARPWLEG LRDHLQFGE PAVVPHPTPY RQVSRVQAKS NPERLRRRLM RRHDLSEEEA RKRIPDTVAR ALDLPFVTLR SQSTGQHFRL F IRHGPLQV TAEEGGFTCY GLSKGGFVPW F |
-分子 #7: CRISPR-associated protein Csy1
| 分子 | 名称: CRISPR-associated protein Csy1 / タイプ: protein_or_peptide / ID: 7 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌) Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌)株: UCBPP-PA14 |
| 分子量 | 理論値: 49.194168 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTSPLPTPTW QELRQFIESF IQERLQGKLD KLQPDEDDKR QTLLATHRRE AWLADAARRV GQLQLVTHTL KPIHPDARGS NLHSLPQAP GQPGLAGSHE LGDRLVSDVV GNAAALDVFK FLSLQYQGKN LLNWLTEDSA EALQALSDNA EQAREWRQAF I GITTVKGA ...文字列: MTSPLPTPTW QELRQFIESF IQERLQGKLD KLQPDEDDKR QTLLATHRRE AWLADAARRV GQLQLVTHTL KPIHPDARGS NLHSLPQAP GQPGLAGSHE LGDRLVSDVV GNAAALDVFK FLSLQYQGKN LLNWLTEDSA EALQALSDNA EQAREWRQAF I GITTVKGA PASHSLAKQL YFPLPGSGYH LLAPLFPTSL VHHVHALLRE ARFGDAAKAA REARSRQESW PHGFSEYPNL AI QKFGGTK PQNISQLNNE RRGENWLLPS LPPNWQRQNV NAPMRHSSVF EHDFGRTPEV SRLTRTLQRF LAKTVHNNLA IRQ RRAQLV AQICDEALQY AARLRELEPG WSATPGCQLH DAEQLWLDPL RAQTDETFLQ RRLRGDWPAE VGNRFANWLN RAVS SDSQI LGSPEAAQWS QELSKELTMF KEILEDERD |
-分子 #4: CRISPR RNA (60-MER)
| 分子 | 名称: CRISPR RNA (60-MER) / タイプ: rna / ID: 4 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa UCBPP-PA14 (緑膿菌) Pseudomonas aeruginosa UCBPP-PA14 (緑膿菌) |
| 分子量 | 理論値: 19.265404 KDa |
| 配列 | 文字列: CUAAGAAAUU CACGGCGGGC UUGAUGUCCG CGUCUACCUG GUUCACUGCC GUGUAGGCAG |
-分子 #5: CRISPR target DNA (44-MER)
| 分子 | 名称: CRISPR target DNA (44-MER) / タイプ: dna / ID: 5 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa UCBPP-PA14 (緑膿菌) Pseudomonas aeruginosa UCBPP-PA14 (緑膿菌) |
| 分子量 | 理論値: 13.584703 KDa |
| 配列 | 文字列: (DC)(DA)(DG)(DG)(DT)(DA)(DG)(DA)(DC)(DG) (DC)(DG)(DG)(DA)(DC)(DA)(DT)(DC)(DA)(DA) (DG)(DC)(DC)(DC)(DG)(DC)(DC)(DG)(DT) (DG)(DA)(DA)(DG)(DG)(DT)(DG)(DC)(DA)(DG) (DC) (DT)(DT)(DC)(DT) |
-分子 #6: Non-complementary R-loop DNA strand
| 分子 | 名称: Non-complementary R-loop DNA strand / タイプ: dna / ID: 6 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa UCBPP-PA14 (緑膿菌) Pseudomonas aeruginosa UCBPP-PA14 (緑膿菌) |
| 分子量 | 理論値: 10.476714 KDa |
| 配列 | 文字列: (DA)(DG)(DA)(DA)(DG)(DC)(DT)(DG)(DC)(DA) (DC)(DC)(DT)(DT)(DC)(DA)(DC)(DG)(DG)(DC) (DG)(DG)(DG)(DC)(DT)(DT)(DG)(DA)(DT) (DG)(DT)(DC)(DC)(DG) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||||||||
| グリッド | モデル: Quantifoil, UltrAuFoil, R1.2/1.3 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 雰囲気: OTHER | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER 詳細: Freezing was carried out in a cold room at 4 degrees C and relative humidity of 98%. 5 uL sample was applied to plasma cleaned grid and manually blotted with Whatman 1 filter paper for 5-7 ...詳細: Freezing was carried out in a cold room at 4 degrees C and relative humidity of 98%. 5 uL sample was applied to plasma cleaned grid and manually blotted with Whatman 1 filter paper for 5-7 sec before plunge freezing.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 温度 | 最低: 77.0 K / 最高: 79.0 K |
| 詳細 | Objective astigmatism was corrected at 36000x magnification using Thon rings visualized with a K2 camera. |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 7676 pixel / デジタル化 - サイズ - 縦: 7420 pixel / デジタル化 - サンプリング間隔: 2.5 µm / デジタル化 - 画像ごとのフレーム数: 1-56 / 撮影したグリッド数: 4 / 実像数: 3208 / 平均露光時間: 14.0 sec. / 平均電子線量: 58.0 e/Å2 詳細: Data were acquired using Leginon and collected on K2 summit operating in super-resolution mode. |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 36000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| 詳細 | The atomic models for Cas5f, Cas8f, Cas6f, and Cas7f from the Csy Acr complex (PDB ID 5UZ9) were used as initial template models for model building. These were individually rigid body-fitted into the reconstructed maps using the fit map function in UCSF Chimera, and residue registers and backbone geometries were fixed in Coot. Models for the crRNA and DNA strands were also manually built into the map using Coot. |
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
| 得られたモデル |  PDB-6ne0: |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)