+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9064 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the PYD filament of AIM2 | |||||||||
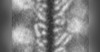
 マップデータ マップデータ | PYD filament of AIM2 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Filament / higher order / innate immunity / inflammasome / IMMUNE SYSTEM | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報pyroptosome complex assembly / AIM2 inflammasome complex assembly / The AIM2 inflammasome / AIM2 inflammasome complex / regulation of behavior / cysteine-type endopeptidase activator activity / Cytosolic sensors of pathogen-associated DNA / pattern recognition receptor signaling pathway / negative regulation of NF-kappaB transcription factor activity / pattern recognition receptor activity ...pyroptosome complex assembly / AIM2 inflammasome complex assembly / The AIM2 inflammasome / AIM2 inflammasome complex / regulation of behavior / cysteine-type endopeptidase activator activity / Cytosolic sensors of pathogen-associated DNA / pattern recognition receptor signaling pathway / negative regulation of NF-kappaB transcription factor activity / pattern recognition receptor activity / pyroptotic inflammatory response / T cell homeostasis / cellular response to interferon-beta / positive regulation of defense response to virus by host / signaling adaptor activity / negative regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / activation of innate immune response / bioluminescence / positive regulation of interleukin-1 beta production / tumor necrosis factor-mediated signaling pathway / generation of precursor metabolites and energy / positive regulation of NF-kappaB transcription factor activity / neuron cellular homeostasis / brain development / cellular response to xenobiotic stimulus / positive regulation of inflammatory response / site of double-strand break / double-stranded DNA binding / defense response to virus / immune response / innate immune response / DNA damage response / mitochondrion / nucleoplasm / identical protein binding / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 5.0 Å | |||||||||
 データ登録者 データ登録者 | Lu A / Li Y | |||||||||
 引用 引用 |  ジャーナル: Cell Discov / 年: 2015 ジャーナル: Cell Discov / 年: 2015タイトル: Plasticity in PYD assembly revealed by cryo-EM structure of the PYD filament of AIM2. 著者: Alvin Lu / Yang Li / Qian Yin / Jianbin Ruan / Xiong Yu / Edward Egelman / Hao Wu /  要旨: Absent in melanoma 2 (AIM2) is an essential cytosolic double-stranded DNA receptor that assembles with the adaptor, apoptosis-associated speck-like protein containing a caspase recruitment domain ...Absent in melanoma 2 (AIM2) is an essential cytosolic double-stranded DNA receptor that assembles with the adaptor, apoptosis-associated speck-like protein containing a caspase recruitment domain (ASC), and caspase-1 to form the AIM2 inflammasome, which leads to proteolytic maturation of cytokines and pyroptotic cell death. AIM2 contains an N-terminal Pyrin domain (PYD) that interacts with ASC through PYD/PYD interactions and nucleates ASC filament formation. To elucidate the molecular basis of AIM2-induced ASC polymerization, we generated AIM2 filaments fused to green fluorescent protein (GFP) and determined its cryo-electron microscopic (cryo-EM) structure. The map showed distinct definition of helices, allowing fitting of the crystal structure. Surprisingly, the GFP-AIM2 filament is a 1-start helix with helical parameters distinct from those of the 3-start ASC filament. However, despite the apparent symmetry difference, helical net and detailed interface analyses reveal minimal changes in subunit packing. GFP-AIM2 nucleated ASC filament formation in comparable efficiency as untagged AIM2, suggesting assembly plasticity in both AIM2 and ASC. The DNA-binding domain of AIM2 is able to form AIM2/DNA filaments, within which the AIM2 is brought into proximity to template ASC filament assembly. Because ASC is able to interact with many PYD-containing receptors for the formation of inflammasomes, the observed structural plasticity may be critically important for this versatility in the PYD/PYD interactions. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9064.map.gz emd_9064.map.gz | 20.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9064-v30.xml emd-9064-v30.xml emd-9064.xml emd-9064.xml | 9.9 KB 9.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_9064.png emd_9064.png | 70.4 KB | ||
| Filedesc metadata |  emd-9064.cif.gz emd-9064.cif.gz | 5.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9064 http://ftp.pdbj.org/pub/emdb/structures/EMD-9064 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9064 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9064 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9064_validation.pdf.gz emd_9064_validation.pdf.gz | 442 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9064_full_validation.pdf.gz emd_9064_full_validation.pdf.gz | 441.6 KB | 表示 | |
| XML形式データ |  emd_9064_validation.xml.gz emd_9064_validation.xml.gz | 5.2 KB | 表示 | |
| CIF形式データ |  emd_9064_validation.cif.gz emd_9064_validation.cif.gz | 5.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9064 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9064 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9064 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9064 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9064.map.gz / 形式: CCP4 / 大きさ: 112.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9064.map.gz / 形式: CCP4 / 大きさ: 112.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | PYD filament of AIM2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.575 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
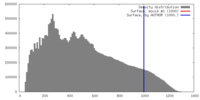
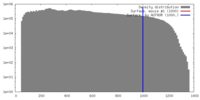
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : PYD of AIM2
| 全体 | 名称: PYD of AIM2 |
|---|---|
| 要素 |
|
-超分子 #1: PYD of AIM2
| 超分子 | 名称: PYD of AIM2 / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Interferon-inducible protein AIM2
| 分子 | 名称: Interferon-inducible protein AIM2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 15 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 10.839629 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: AMESKYKEIL LLTGLDNITD EELDRFKGFL SDEFNIATGK LHTANRIQVA TLMIQNAGAV SAVMKTIRIF QKLNYMLLAK RLQEEKEKV DKQYK UniProtKB: Interferon-inducible protein AIM2 |
-分子 #2: Green fluorescent protein
| 分子 | 名称: Green fluorescent protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 15 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 25.924553 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SKGEELFTGV VPILVELDGD VNGHKFSVSG EGEGDATYGK LTLKFICTTG KLPVPWPTLV TTF(CRO)VQCFSR YPDH (MSE)KRHD FFKSA(MSE)PEGY VQERTIFFKD DGNYKTRAEV KFEGDTLVNR IELKGIDFKE DGNILGHKLE YNYNSHN VY I(MSE) ...文字列: SKGEELFTGV VPILVELDGD VNGHKFSVSG EGEGDATYGK LTLKFICTTG KLPVPWPTLV TTF(CRO)VQCFSR YPDH (MSE)KRHD FFKSA(MSE)PEGY VQERTIFFKD DGNYKTRAEV KFEGDTLVNR IELKGIDFKE DGNILGHKLE YNYNSHN VY I(MSE)ADKQKNGI KVNFKIRHNI EDGSVQLADH YQQNTPIGDG PVLLPDNHYL STQSALSKDP NEKRDH(MSE)VLL EFVTAAGI UniProtKB: Green fluorescent protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 6.0 Å 想定した対称性 - らせんパラメータ - ΔΦ: 138.9 ° 想定した対称性 - らせんパラメータ - 軸対称性: C1 (非対称) 解像度のタイプ: BY AUTHOR / 解像度: 5.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 54973 |
|---|---|
| 初期モデル | モデルのタイプ: OTHER / 詳細: Solid cylinder |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)