+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9013 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | EsCas13d-crRNA binary complex | |||||||||
 マップデータ マップデータ | EsCas13d-crRNA binary complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CRISPR-Cas / RNase / Complex / RNA BINDING PROTEIN-RNA complex | |||||||||
| 機能・相同性 | Uncharacterized protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  unidentified bacterium (バクテリア) / unidentified bacterium (バクテリア) /  bacterium (バクテリア) / bacterium (バクテリア) /  [Eubacterium] siraeum DSM 15702 (バクテリア) [Eubacterium] siraeum DSM 15702 (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Zhang C / Lyumkis D | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2018 ジャーナル: Cell / 年: 2018タイトル: Structural Basis for the RNA-Guided Ribonuclease Activity of CRISPR-Cas13d. 著者: Cheng Zhang / Silvana Konermann / Nicholas J Brideau / Peter Lotfy / Xuebing Wu / Scott J Novick / Timothy Strutzenberg / Patrick R Griffin / Patrick D Hsu / Dmitry Lyumkis /  要旨: CRISPR-Cas endonucleases directed against foreign nucleic acids mediate prokaryotic adaptive immunity and have been tailored for broad genetic engineering applications. Type VI-D CRISPR systems ...CRISPR-Cas endonucleases directed against foreign nucleic acids mediate prokaryotic adaptive immunity and have been tailored for broad genetic engineering applications. Type VI-D CRISPR systems contain the smallest known family of single effector Cas enzymes, and their signature Cas13d ribonuclease employs guide RNAs to cleave matching target RNAs. To understand the molecular basis for Cas13d function and explain its compact molecular architecture, we resolved cryoelectron microscopy structures of Cas13d-guide RNA binary complex and Cas13d-guide-target RNA ternary complex to 3.4 and 3.3 Å resolution, respectively. Furthermore, a 6.5 Å reconstruction of apo Cas13d combined with hydrogen-deuterium exchange revealed conformational dynamics that have implications for RNA scanning. These structures, together with biochemical and cellular characterization, provide insights into its RNA-guided, RNA-targeting mechanism and delineate a blueprint for the rational design of improved transcriptome engineering technologies. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9013.map.gz emd_9013.map.gz | 56.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9013-v30.xml emd-9013-v30.xml emd-9013.xml emd-9013.xml | 22.2 KB 22.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_9013.png emd_9013.png | 173.9 KB | ||
| Filedesc metadata |  emd-9013.cif.gz emd-9013.cif.gz | 6.9 KB | ||
| その他 |  emd_9013_additional.map.gz emd_9013_additional.map.gz emd_9013_half_map_1.map.gz emd_9013_half_map_1.map.gz emd_9013_half_map_2.map.gz emd_9013_half_map_2.map.gz | 5.8 MB 11.8 MB 11.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9013 http://ftp.pdbj.org/pub/emdb/structures/EMD-9013 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9013 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9013 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9013_validation.pdf.gz emd_9013_validation.pdf.gz | 799.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9013_full_validation.pdf.gz emd_9013_full_validation.pdf.gz | 798.7 KB | 表示 | |
| XML形式データ |  emd_9013_validation.xml.gz emd_9013_validation.xml.gz | 12.4 KB | 表示 | |
| CIF形式データ |  emd_9013_validation.cif.gz emd_9013_validation.cif.gz | 14.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9013 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9013 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9013 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9013 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9013.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9013.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EsCas13d-crRNA binary complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.79 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: 3D FSC map
| ファイル | emd_9013_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D FSC map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
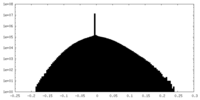
| 密度ヒストグラム |
-ハーフマップ: EsCas13d-crRNA binary complex, half map #1
| ファイル | emd_9013_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EsCas13d-crRNA binary complex, half map #1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
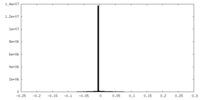
| 密度ヒストグラム |
-ハーフマップ: EsCas13d-crRNA binary complex, half map #2
| ファイル | emd_9013_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EsCas13d-crRNA binary complex, half map #2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
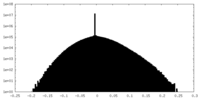
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Binary complex of EsCas13d with crRNA and Mg2+
| 全体 | 名称: Binary complex of EsCas13d with crRNA and Mg2+ |
|---|---|
| 要素 |
|
-超分子 #1: Binary complex of EsCas13d with crRNA and Mg2+
| 超分子 | 名称: Binary complex of EsCas13d with crRNA and Mg2+ / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  unidentified bacterium (バクテリア) unidentified bacterium (バクテリア) |
| 分子量 | 理論値: 125 KDa |
-分子 #1: crRNA (52-MER)
| 分子 | 名称: crRNA (52-MER) / タイプ: rna / ID: 1 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  bacterium (バクテリア) bacterium (バクテリア) |
| 分子量 | 理論値: 16.385846 KDa |
| 配列 | 文字列: CACCCGUGCA AAAAUGCAGG GGUCUAAAAC GACCUGAAUA UUUCAGAUCA A |
-分子 #2: EsCas13d
| 分子 | 名称: EsCas13d / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  [Eubacterium] siraeum DSM 15702 (バクテリア) [Eubacterium] siraeum DSM 15702 (バクテリア) |
| 分子量 | 理論値: 110.828938 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGKKIHARDL REQRKTDRTE KFADQNKKRE AERAVPKKDA AVSVKSVSSV SSKKDNVTKS MAKAAGVKSV FAVGNTVYMT SFGRGNDAV LEQKIVDTSH EPLNIDDPAY QLNVVTMNGY SVTGHRGETV SAVTDNPLRR FNGRKKDEPE QSVPTDMLCL K PTLEKKFF ...文字列: MGKKIHARDL REQRKTDRTE KFADQNKKRE AERAVPKKDA AVSVKSVSSV SSKKDNVTKS MAKAAGVKSV FAVGNTVYMT SFGRGNDAV LEQKIVDTSH EPLNIDDPAY QLNVVTMNGY SVTGHRGETV SAVTDNPLRR FNGRKKDEPE QSVPTDMLCL K PTLEKKFF GKEFDDNIHI QLIYNILDIE KILAVYSTNA IYALNNMSAD ENIENSDFFM KRTTDETFDD FEKKKESTNS RE KADFDAF EKFIGNYRLA YFADAFYVNK KNPKGKAKNV LREDKELYSV LTLIGKLRHW CVHSEEGRAE FWLYKLDELK DDF KNVLDV VYNRPVEEIN NRFIENNKVN IQILGSVYKN TDIAELVRSY YEFLITKKYK NMGFSIKKLR ESMLEGKGYA DKEY DSVRN KLYQMTDFIL YTGYINEDSD RADDLVNTLR SSLKEDDKTT VYCKEADYLW KKYRESIREV ADALDGDNIK KLSKS NIEI QEDKLRKCFI SYADSVSEFT KLIYLLTRFL SGKEINDLVT TLINKFDNIR SFLEIMDELG LDRTFTAEYS FFEGST KYL AELVELNSFV KSCSFDINAK RTMYRDALDI LGIESDKTEE DIEKMIDNIL QIDANGDKKL KKNNGLRNFI ASNVIDS NR FKYLVRYGNP KKIRETAKCK PAVRFVLNEI PDAQIERYYE ACCPKNTALC SANKRREKLA DMIAEIKFEN FSDAGNYQ K ANVTSRTSEA EIKRKNQAII RLYLTVMYIM LKNLVNVNAR YVIAFHCVER DTKLYAESGL EVGNIEKNKT NLTMAVMGV KLENGIIKTE FDKSFAENAA NRYLRNARWY KLILDNLKKS ERAVVNEFRN TVCHLNAIRN ININIKEIKE VENYFALYHY LIQKHLENR FADKKVERDT GDFISKLEEH KTYCKDFVKA YCTPFGYNLV RYKNLTIDGL FDKNYPGKDD SDEQK UniProtKB: Uncharacterized protein |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||||||||
| グリッド | モデル: Quantifoil, UltrAuFoil, R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 7 sec. / 前処理 - 雰囲気: OTHER | |||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 80 % / 装置: HOMEMADE PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-120 / 実像数: 1435 / 平均電子線量: 56.8 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 57000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: FOURIER SPACE / 解像度のタイプ: BY AUTHOR / 解像度: 3.4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cisTEM / 使用した粒子像数: 43786 |
| 初期 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: RELION |
| 最終 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: cisTEM |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 当てはまり具合の基準: Correlation coefficient |
|---|---|
| 得られたモデル |  PDB-6e9e: |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)