+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8951 | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | PTEX Core Complex in the Engaged (Extended) State | ||||||||||||||||||||||||||||||||||||
 マップデータ マップデータ | PTEX Core Complex in the Engaged (Extended) State | ||||||||||||||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||||||||||||||
 キーワード キーワード | Translocon / Membrane Protein / ATPase / PROTEIN TRANSPORT | ||||||||||||||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報PTEX complex / apical complex / symbiont-containing vacuole / translocation of peptides or proteins into host cell cytoplasm / symbiont-containing vacuole membrane / response to unfolded protein / cellular response to heat / response to heat / ATP hydrolysis activity / ATP binding / cytoplasm 類似検索 - 分子機能 | ||||||||||||||||||||||||||||||||||||
| 生物種 |   | ||||||||||||||||||||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.16 Å | ||||||||||||||||||||||||||||||||||||
 データ登録者 データ登録者 | Ho C / Lai M | ||||||||||||||||||||||||||||||||||||
| 資金援助 |  米国, 11件 米国, 11件
| ||||||||||||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2018 ジャーナル: Nature / 年: 2018タイトル: Malaria parasite translocon structure and mechanism of effector export. 著者: Chi-Min Ho / Josh R Beck / Mason Lai / Yanxiang Cui / Daniel E Goldberg / Pascal F Egea / Z Hong Zhou /  要旨: The putative Plasmodium translocon of exported proteins (PTEX) is essential for transport of malarial effector proteins across a parasite-encasing vacuolar membrane into host erythrocytes, but the ...The putative Plasmodium translocon of exported proteins (PTEX) is essential for transport of malarial effector proteins across a parasite-encasing vacuolar membrane into host erythrocytes, but the mechanism of this process remains unknown. Here we show that PTEX is a bona fide translocon by determining structures of the PTEX core complex at near-atomic resolution using cryo-electron microscopy. We isolated the endogenous PTEX core complex containing EXP2, PTEX150 and HSP101 from Plasmodium falciparum in the 'engaged' and 'resetting' states of endogenous cargo translocation using epitope tags inserted using the CRISPR-Cas9 system. In the structures, EXP2 and PTEX150 interdigitate to form a static, funnel-shaped pseudo-seven-fold-symmetric protein-conducting channel spanning the vacuolar membrane. The spiral-shaped AAA+ HSP101 hexamer is tethered above this funnel, and undergoes pronounced compaction that allows three of six tyrosine-bearing pore loops lining the HSP101 channel to dissociate from the cargo, resetting the translocon for the next threading cycle. Our work reveals the mechanism of P. falciparum effector export, and will inform structure-based design of drugs targeting this unique translocon. | ||||||||||||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8951.map.gz emd_8951.map.gz | 27 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8951-v30.xml emd-8951-v30.xml emd-8951.xml emd-8951.xml | 25.2 KB 25.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8951.png emd_8951.png | 164.2 KB | ||
| Filedesc metadata |  emd-8951.cif.gz emd-8951.cif.gz | 7.2 KB | ||
| その他 |  emd_8951_additional_1.map.gz emd_8951_additional_1.map.gz emd_8951_additional_2.map.gz emd_8951_additional_2.map.gz | 24.4 MB 22.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8951 http://ftp.pdbj.org/pub/emdb/structures/EMD-8951 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8951 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8951 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8951_validation.pdf.gz emd_8951_validation.pdf.gz | 370.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8951_full_validation.pdf.gz emd_8951_full_validation.pdf.gz | 370.4 KB | 表示 | |
| XML形式データ |  emd_8951_validation.xml.gz emd_8951_validation.xml.gz | 7.7 KB | 表示 | |
| CIF形式データ |  emd_8951_validation.cif.gz emd_8951_validation.cif.gz | 8.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8951 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8951 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8951 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8951 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8951.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8951.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | PTEX Core Complex in the Engaged (Extended) State | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: PTEX Core Complex in the Engaged (Extended) State, additional map #1
| ファイル | emd_8951_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | PTEX Core Complex in the Engaged (Extended) State, additional map #1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
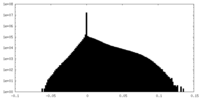
| 密度ヒストグラム |
-追加マップ: PTEX Core Complex in the Engaged (Extended) State, additional map #2
| ファイル | emd_8951_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | PTEX Core Complex in the Engaged (Extended) State, additional map #2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
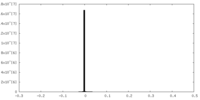
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Plasmodium Translocon of Exported Proteins (PTEX) Core Complex
| 全体 | 名称: Plasmodium Translocon of Exported Proteins (PTEX) Core Complex |
|---|---|
| 要素 |
|
-超分子 #1: Plasmodium Translocon of Exported Proteins (PTEX) Core Complex
| 超分子 | 名称: Plasmodium Translocon of Exported Proteins (PTEX) Core Complex タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Heat shock protein 101
| 分子 | 名称: Heat shock protein 101 / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 106.277125 KDa |
| 配列 | 文字列: MTRRYLKYYI FVTLLFFVQV INNVLCAPDN KQEQGKYLNR TINILNAGKN IAKSYGHNKL KPIHILSALA KSDYGSTLFK ENNVNAANL KEYIDIALEQ TRAGAPLDNK SKIVNSAEVK ETLALAEAAA NKYKSPKVDV EHLLSGLSND ELVNEIFNEV Y LTDEAIKA ...文字列: MTRRYLKYYI FVTLLFFVQV INNVLCAPDN KQEQGKYLNR TINILNAGKN IAKSYGHNKL KPIHILSALA KSDYGSTLFK ENNVNAANL KEYIDIALEQ TRAGAPLDNK SKIVNSAEVK ETLALAEAAA NKYKSPKVDV EHLLSGLSND ELVNEIFNEV Y LTDEAIKA ILKRKFEKTK KDKDGKTGTL YIEQFGSNMN EKVRNGKLQG IYGRDEEIRA IIESLLRYNK NSPVLVGNPG TG KTTIVEG LVYRIEKGDV PKELQGYTVI SLNFRKFTSG TSYRGEFETR MKNIIKELKN KKNKIILFVD EIHLLLGAGK AEG GTDAAN LLKPVLSKGE IKLIGATTIA EYRKFIESCS AFERRFEKIL VEPPSVDMTV KILRSLKSKY ENFYGINITD KALV AAAKI SDRFIKDRYL PDKAIDLLNK ACSFLQVQLS GKPRIIDVTE RDIERLSYEI STLEKDVDKV SKKKYNKLIK EFEEK KEQL KKYYEEYVIT GERLKRKKEI EKKLNDLKEL TQNYVYSNKE PPIELQNSLK EAQQKYLELY KETVAYVEAK THNAMN VDA VYQEHVSYIY LRDSGMPLGS LSFESSKGAL KLYNSLSKSI IGNEDIIKSL SDAVVKAATG MKDPEKPIGT FLFLGPT GV GKTELAKTLA IELFNSKDNL IRVNMSEFTE AHSVSKITGS PPGYVGFSDS GQLTEAVREK PHSVVLFDEL EKAHADVF K VLLQILGDGY INDNHRRNID FSNTIIIMTS NLGAELFKKK LFFDADNSGT PEYKRVMEDV RLSLIKKCKK VFKPEFVNR IDKIGVFEPL NKKNLHKIVA LRFKKLEKRL EEKNIQVSVS EKAIDYIIDQ SYDPELGARP TLIFIESVIM TKFAIMYLKK ELVDDMDVF VDYNSKAKNL VINLSKTPRD YKDDDDKDYK DDDDKDYKDD DDK UniProtKB: Heat shock protein 101 |
-分子 #2: Exported protein 2
| 分子 | 名称: Exported protein 2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 33.458707 KDa |
| 配列 | 文字列: MKVSYIFSFF LLFFVYKNTN TVVCDNGYGD LAATSALTTV IKDPISLTIK DIYEHGVKNP FTKIIHKLKK FIRYRKVLRW SRMWWVLLV REIVGDNTIE KKTEKALREI WDQCTIAVYN NTLNAVESKP LLFLHGILNE CRNNFATKLR QDPSLIVAKI D QIIKSQIY ...文字列: MKVSYIFSFF LLFFVYKNTN TVVCDNGYGD LAATSALTTV IKDPISLTIK DIYEHGVKNP FTKIIHKLKK FIRYRKVLRW SRMWWVLLV REIVGDNTIE KKTEKALREI WDQCTIAVYN NTLNAVESKP LLFLHGILNE CRNNFATKLR QDPSLIVAKI D QIIKSQIY RFWVSEPYLK IGRSHTLYTH ITPDAVPQLP KECTLKHLSS YMEEKLKSME SKKNIESGKY EFDVDSSETD ST KDDGKPD DDDDDDDNFD DDDNFDDDTV EEEDASGDLF KNEKKDENKE UniProtKB: Exported protein 2 |
-分子 #3: Translocon component PTEX150
| 分子 | 名称: Translocon component PTEX150 / タイプ: protein_or_peptide / ID: 3 / コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 22.59317 KDa |
| 配列 | 文字列: SVKDIKKLIE EGILDYEDLT ENELRKLAKP DDNFYELSPY ASDEKDLSLN ETSGLTNEQL KNFLGQNGTY HMSYDSKSID YAKQKKSEK KEDQQEDDDG FYDAYKQIKN SYDGIPNNFN HEAPQLIGNN YVFTSIYDTK ENLIKFLKKN SEYDLYD (UNK)(UNK) (UNK) ...文字列: SVKDIKKLIE EGILDYEDLT ENELRKLAKP DDNFYELSPY ASDEKDLSLN ETSGLTNEQL KNFLGQNGTY HMSYDSKSID YAKQKKSEK KEDQQEDDDG FYDAYKQIKN SYDGIPNNFN HEAPQLIGNN YVFTSIYDTK ENLIKFLKKN SEYDLYD (UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK) (UNK)(UNK) UniProtKB: Translocon component PTEX150 |
-分子 #4: Endogenous cargo polypeptide
| 分子 | 名称: Endogenous cargo polypeptide / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 1.294587 KDa |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK) |
-分子 #5: Unknown (Claw)
| 分子 | 名称: Unknown (Claw) / タイプ: protein_or_peptide / ID: 5 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 4.954098 KDa |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) ...文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK) |
-分子 #6: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 6 / コピー数: 12 / 式: AGS |
|---|---|
| 分子量 | 理論値: 523.247 Da |
| Chemical component information |  ChemComp-AGS: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: LACEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
| 詳細 | PTEX core complex purified from P. falciparum parasites cultured in human erythrocytes |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3710 pixel / デジタル化 - サイズ - 縦: 3838 pixel / デジタル化 - 画像ごとのフレーム数: 2-50 / 平均露光時間: 10.0 sec. / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 4.0 µm / 最小 デフォーカス(補正後): 1.5 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-6e10: |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)