+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8911 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of a mitochondrial calcium uniporter | |||||||||
 マップデータ マップデータ | Single-particle cryo-EM reconstruction of the N. crassa mitochondrial calcium uniporter | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | mitochondrial calcium uniporter / calcium-selective ion channel / calcium uptake / uniporter / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報uniporter activity / uniplex complex / mitochondrial calcium ion homeostasis / calcium import into the mitochondrion / calcium channel activity / protein homotetramerization / mitochondrial inner membrane / metal ion binding / identical protein binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Neurospora crassa (菌類) Neurospora crassa (菌類) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.7 Å | |||||||||
 データ登録者 データ登録者 | Yoo J / Wu M | |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2018 ジャーナル: Science / 年: 2018タイトル: Cryo-EM structure of a mitochondrial calcium uniporter. 著者: Jiho Yoo / Mengyu Wu / Ying Yin / Mark A Herzik / Gabriel C Lander / Seok-Yong Lee /  要旨: Calcium transport plays an important role in regulating mitochondrial physiology and pathophysiology. The mitochondrial calcium uniporter (MCU) is a calcium-selective ion channel that is the primary ...Calcium transport plays an important role in regulating mitochondrial physiology and pathophysiology. The mitochondrial calcium uniporter (MCU) is a calcium-selective ion channel that is the primary mediator for calcium uptake into the mitochondrial matrix. Here, we present the cryo-electron microscopy structure of the full-length MCU from to an overall resolution of ~3.7 angstroms. Our structure reveals a tetrameric architecture, with the soluble and transmembrane domains adopting different symmetric arrangements within the channel. The conserved W-D-Φ-Φ-E-P-V-T-Y sequence motif of MCU pore forms a selectivity filter comprising two acidic rings separated by one helical turn along the central axis of the channel pore. The structure combined with mutagenesis gives insight into the basis of calcium recognition. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8911.map.gz emd_8911.map.gz | 59.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8911-v30.xml emd-8911-v30.xml emd-8911.xml emd-8911.xml | 16.8 KB 16.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_8911_fsc.xml emd_8911_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_8911.png emd_8911.png | 155.8 KB | ||
| Filedesc metadata |  emd-8911.cif.gz emd-8911.cif.gz | 5.7 KB | ||
| その他 |  emd_8911_half_map_1.map.gz emd_8911_half_map_1.map.gz emd_8911_half_map_2.map.gz emd_8911_half_map_2.map.gz | 48.3 MB 48.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8911 http://ftp.pdbj.org/pub/emdb/structures/EMD-8911 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8911 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8911 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8911_validation.pdf.gz emd_8911_validation.pdf.gz | 883.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8911_full_validation.pdf.gz emd_8911_full_validation.pdf.gz | 883.2 KB | 表示 | |
| XML形式データ |  emd_8911_validation.xml.gz emd_8911_validation.xml.gz | 16 KB | 表示 | |
| CIF形式データ |  emd_8911_validation.cif.gz emd_8911_validation.cif.gz | 20.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8911 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8911 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8911 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8911 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8911.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8911.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Single-particle cryo-EM reconstruction of the N. crassa mitochondrial calcium uniporter | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.15 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: N. crassa mitochondrial calcium uniporter, half map 1
| ファイル | emd_8911_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | N. crassa mitochondrial calcium uniporter, half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
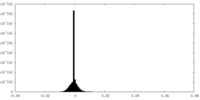
| 密度ヒストグラム |
-ハーフマップ: N. crassa mitochondrial calcium uniporter, half map 2
| ファイル | emd_8911_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | N. crassa mitochondrial calcium uniporter, half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
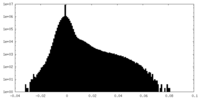
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Mitochondrial Calcium Uniporter
| 全体 | 名称: Mitochondrial Calcium Uniporter |
|---|---|
| 要素 |
|
-超分子 #1: Mitochondrial Calcium Uniporter
| 超分子 | 名称: Mitochondrial Calcium Uniporter / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Neurospora crassa (菌類) Neurospora crassa (菌類) |
-分子 #1: Mitochondrial calcium uniporter
| 分子 | 名称: Mitochondrial calcium uniporter / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Neurospora crassa (菌類) Neurospora crassa (菌類) |
| 分子量 | 理論値: 52.948117 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MASTSVSPKT RETEAEAKAK KLDQKRLDEH EEEVRAREQQ VRRPWHREGA DKPPVEGNAD PIAKGKLLTT PTRLLKLILP LPLRVEKDQ KNNGRNNEYG RSISLNSDIQ PLALLIHPQQ PLSYVERLIQ AELPPVVENG QEKIPNVYFR AEDSEQGDQK P TSRAEARS ...文字列: MASTSVSPKT RETEAEAKAK KLDQKRLDEH EEEVRAREQQ VRRPWHREGA DKPPVEGNAD PIAKGKLLTT PTRLLKLILP LPLRVEKDQ KNNGRNNEYG RSISLNSDIQ PLALLIHPQQ PLSYVERLIQ AELPPVVENG QEKIPNVYFR AEDSEQGDQK P TSRAEARS KDDGGEPSEY NTNLSHVASA SGLGHRGPKR SSQDKRWVRW SSSTEMGDFI RDAARGREFA IEIEGYNIEM RV SVPSFGD RTYYMRQRLR KMSSEIDGLA KIKHECDLLA HRSAHRLAKG GFGLLAGWWG VVYYVTFHTE FGWDLVEPVT YLA GLTTIM GGYLWFLYIN KDLSYKAAMN VTVSRRQHAL YEMKGFDIER WEQLVQDANA LRREIRVIAV EYDVDWDETR DVGE DVKDV LDEERSRRDD EHRSIEKEKD EKFTEDEKRK RKKDKESKET SGDSTNSHHH HHH UniProtKB: Calcium uniporter protein, mitochondrial |
-分子 #2: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 2 / コピー数: 1 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.2 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil, UltrAuFoil, R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 7 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: HOMEMADE PLUNGER / 詳細: 4 second blot time. |
| 詳細 | Final reconstruction comprises ~80% of MCU in nanodisc (crosslinked using BS3) and ~20% MCU in amphipol. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3710 pixel / デジタル化 - サイズ - 縦: 3838 pixel / 平均電子線量: 65.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 36000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 温度因子: 100 |
|---|---|
| 得られたモデル |  PDB-6dt0: |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)