+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8903 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structures of ASC and NLRC4 CARD Filaments Reveal a Unified Mechanism of Nucleation and Activation of Caspase-1 | |||||||||
 マップデータ マップデータ | Helical reconstruction of NLRC4-CARD | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報IPAF inflammasome complex / The IPAF inflammasome / icosanoid biosynthetic process / canonical inflammasome complex / caspase binding / positive regulation of protein processing / pattern recognition receptor signaling pathway / TP53 Regulates Transcription of Caspase Activators and Caspases / pyroptotic inflammatory response / detection of bacterium ...IPAF inflammasome complex / The IPAF inflammasome / icosanoid biosynthetic process / canonical inflammasome complex / caspase binding / positive regulation of protein processing / pattern recognition receptor signaling pathway / TP53 Regulates Transcription of Caspase Activators and Caspases / pyroptotic inflammatory response / detection of bacterium / endopeptidase activator activity / activation of innate immune response / positive regulation of interleukin-1 beta production / : / protein homooligomerization / positive regulation of inflammatory response / defense response to bacterium / positive regulation of apoptotic process / inflammatory response / innate immune response / intracellular membrane-bounded organelle / apoptotic process / magnesium ion binding / protein homodimerization activity / ATP binding / identical protein binding / plasma membrane / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.58 Å | |||||||||
 データ登録者 データ登録者 | Li Y / Fu T / Wu H | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2018 ジャーナル: Proc Natl Acad Sci U S A / 年: 2018タイトル: Cryo-EM structures of ASC and NLRC4 CARD filaments reveal a unified mechanism of nucleation and activation of caspase-1. 著者: Yang Li / Tian-Min Fu / Alvin Lu / Kristen Witt / Jianbin Ruan / Chen Shen / Hao Wu /  要旨: Canonical inflammasomes are cytosolic supramolecular complexes that activate caspase-1 upon sensing extrinsic microbial invasions and intrinsic sterile stress signals. During inflammasome assembly, ...Canonical inflammasomes are cytosolic supramolecular complexes that activate caspase-1 upon sensing extrinsic microbial invasions and intrinsic sterile stress signals. During inflammasome assembly, adaptor proteins ASC and NLRC4 recruit caspase-1 through homotypic caspase recruitment domain (CARD) interactions, leading to caspase-1 dimerization and activation. Activated caspase-1 processes proinflammatory cytokines and Gasdermin D to induce cytokine maturation and pyroptotic cell death. Here, we present cryo-electron microscopy (cryo-EM) structures of NLRC4 CARD and ASC CARD filaments mediated by conserved three types of asymmetric interactions (types I, II, and III). We find that the CARDs of these two adaptor proteins share a similar assembly pattern, which matches that of the caspase-1 CARD filament whose structure we defined previously. These data indicate a unified mechanism for downstream caspase-1 recruitment through CARD-CARD interactions by both adaptors. Using structure modeling, we further show that full-length NLRC4 assembles via two separate symmetries at its CARD and its nucleotide-binding domain (NBD), respectively. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8903.map.gz emd_8903.map.gz | 42.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8903-v30.xml emd-8903-v30.xml emd-8903.xml emd-8903.xml | 9.6 KB 9.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8903.png emd_8903.png | 78.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8903 http://ftp.pdbj.org/pub/emdb/structures/EMD-8903 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8903 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8903 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8903_validation.pdf.gz emd_8903_validation.pdf.gz | 533.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8903_full_validation.pdf.gz emd_8903_full_validation.pdf.gz | 533.4 KB | 表示 | |
| XML形式データ |  emd_8903_validation.xml.gz emd_8903_validation.xml.gz | 6.1 KB | 表示 | |
| CIF形式データ |  emd_8903_validation.cif.gz emd_8903_validation.cif.gz | 6.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8903 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8903 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8903 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8903 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8903.map.gz / 形式: CCP4 / 大きさ: 45.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8903.map.gz / 形式: CCP4 / 大きさ: 45.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Helical reconstruction of NLRC4-CARD | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.32 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
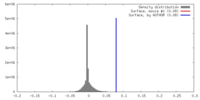
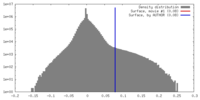
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Caspase recruitment domain of NLR family CARD domain-containing p...
| 全体 | 名称: Caspase recruitment domain of NLR family CARD domain-containing protein 4 |
|---|---|
| 要素 |
|
-超分子 #1: Caspase recruitment domain of NLR family CARD domain-containing p...
| 超分子 | 名称: Caspase recruitment domain of NLR family CARD domain-containing protein 4 タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種: |
-分子 #1: NLR family CARD domain-containing protein 4
| 分子 | 名称: NLR family CARD domain-containing protein 4 / タイプ: protein_or_peptide / ID: 1 / コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 9.987671 KDa |
| 組換発現 | 生物種: |
| 配列 | 文字列: MNFIKDNSRA LIQRMGMTVI KQITDDLFVW NVLNREEVNI ICCEKVEQDA ARGIIHMILK KGSESCNLFL KSLKEWNYPL FQDLN |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 41.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 4.93 Å 想定した対称性 - らせんパラメータ - ΔΦ: -100.48 ° 想定した対称性 - らせんパラメータ - 軸対称性: C1 (非対称) 解像度のタイプ: BY AUTHOR / 解像度: 3.58 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 199312 |
|---|---|
| 最終 角度割当 | タイプ: NOT APPLICABLE |
 ムービー
ムービー コントローラー
コントローラー
























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)