+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8302 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | HIV Clade B Env SOSIP ectodomain trimer from the Amsterdam Cohort Study patient AMC011 in complex with Fab from IgG PGV04 | |||||||||
 マップデータ マップデータ | HIV Clade B Env SOSIP ectodomain trimer from the Amsterdam Cohort Study patient AMC011 in complex with Fab from IgG PGV04 | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) | |||||||||
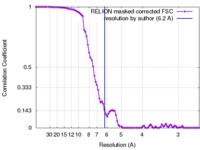
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.2 Å | |||||||||
 データ登録者 データ登録者 | Pallesen J / de Val N / Ozorowski G / Cottrell CA / Ward AB | |||||||||
 引用 引用 |  ジャーナル: Nat Microbiol / 年: 2016 ジャーナル: Nat Microbiol / 年: 2016タイトル: An HIV-1 antibody from an elite neutralizer implicates the fusion peptide as a site of vulnerability. 著者: Marit J van Gils / Tom L G M van den Kerkhof / Gabriel Ozorowski / Christopher A Cottrell / Devin Sok / Matthias Pauthner / Jesper Pallesen / Natalia de Val / Anila Yasmeen / Steven W de ...著者: Marit J van Gils / Tom L G M van den Kerkhof / Gabriel Ozorowski / Christopher A Cottrell / Devin Sok / Matthias Pauthner / Jesper Pallesen / Natalia de Val / Anila Yasmeen / Steven W de Taeye / Anna Schorcht / Stephanie Gumbs / Inez Johanna / Karen Saye-Francisco / Chi-Hui Liang / Elise Landais / Xiaoyan Nie / Laura K Pritchard / Max Crispin / Garnett Kelsoe / Ian A Wilson / Hanneke Schuitemaker / Per Johan Klasse / John P Moore / Dennis R Burton / Andrew B Ward / Rogier W Sanders /    要旨: The induction by vaccination of broadly neutralizing antibodies (bNAbs) capable of neutralizing various HIV-1 viral strains is challenging, but understanding how a subset of HIV-infected individuals ...The induction by vaccination of broadly neutralizing antibodies (bNAbs) capable of neutralizing various HIV-1 viral strains is challenging, but understanding how a subset of HIV-infected individuals develops bNAbs may guide immunization strategies. Here, we describe the isolation and characterization of the bNAb ACS202 from an elite neutralizer that recognizes a new, trimer-specific and cleavage-dependent epitope at the gp120-gp41 interface of the envelope glycoprotein (Env), involving the glycan N88 and the gp41 fusion peptide. In addition, an Env trimer, AMC011 SOSIP.v4.2, based on early virus isolates from the same elite neutralizer, was constructed, and its structure by cryo-electron microscopy at 6.2 Å resolution reveals a closed, pre-fusion conformation similar to that of the BG505 SOSIP.664 trimer. The availability of a native-like Env trimer and a bNAb from the same elite neutralizer provides the opportunity to design vaccination strategies aimed at generating similar bNAbs against a key functional site on HIV-1. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8302.map.gz emd_8302.map.gz | 59.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8302-v30.xml emd-8302-v30.xml emd-8302.xml emd-8302.xml | 23.4 KB 23.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_8302_fsc.xml emd_8302_fsc.xml | 8.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_8302.png emd_8302.png | 172.8 KB | ||
| その他 |  emd_8302_additional.map.gz emd_8302_additional.map.gz emd_8302_half_map_1.map.gz emd_8302_half_map_1.map.gz emd_8302_half_map_2.map.gz emd_8302_half_map_2.map.gz | 59.7 MB 49.6 MB 49.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8302 http://ftp.pdbj.org/pub/emdb/structures/EMD-8302 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8302 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8302 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8302_validation.pdf.gz emd_8302_validation.pdf.gz | 78.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8302_full_validation.pdf.gz emd_8302_full_validation.pdf.gz | 77.7 KB | 表示 | |
| XML形式データ |  emd_8302_validation.xml.gz emd_8302_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8302 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8302 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8302 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8302 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8302.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8302.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | HIV Clade B Env SOSIP ectodomain trimer from the Amsterdam Cohort Study patient AMC011 in complex with Fab from IgG PGV04 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.31 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Additional map: HIV Clade B Env SOSIP ectodomain...
| ファイル | emd_8302_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Additional map: HIV Clade B Env SOSIP ectodomain trimer from the Amsterdam Cohort Study patient AMC011 in complex with Fab from IgG PGV04 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map #1: HIV Clade B Env SOSIP...
| ファイル | emd_8302_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map #1: HIV Clade B Env SOSIP ectodomain trimer from the Amsterdam Cohort Study patient AMC011 in complex with Fab from IgG PGV04 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map #2: HIV Clade B Env SOSIP...
| ファイル | emd_8302_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map #2: HIV Clade B Env SOSIP ectodomain trimer from the Amsterdam Cohort Study patient AMC011 in complex with Fab from IgG PGV04 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : HIV Clade B Env SOSIP ectodomain trimer from the Amsterdam Cohort...
| 全体 | 名称: HIV Clade B Env SOSIP ectodomain trimer from the Amsterdam Cohort Study patient AMC011 in complex with Fab from IgG PGV04 |
|---|---|
| 要素 |
|
-超分子 #1: HIV Clade B Env SOSIP ectodomain trimer from the Amsterdam Cohort...
| 超分子 | 名称: HIV Clade B Env SOSIP ectodomain trimer from the Amsterdam Cohort Study patient AMC011 in complex with Fab from IgG PGV04 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 分子量 | 理論値: 600 KDa |
-分子 #1: PGV04 Fab HC
| 分子 | 名称: PGV04 Fab HC / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQLVQSGSG VKKPGASVRV SCWTSEDIFE RTELIHWVRQ APGQGLEWIG WVKTVTGAVN FGSPDFRQRV SLTRDRDLFT AHMDIRGLTQ GDTATYFCAR QKFYTGGQGW YFDLWGRGTL IVVSSASTKG PSVFPLAPSS KSTSGGTAAL GCLVKDYFPE PVTVSWNSGA ...文字列: QVQLVQSGSG VKKPGASVRV SCWTSEDIFE RTELIHWVRQ APGQGLEWIG WVKTVTGAVN FGSPDFRQRV SLTRDRDLFT AHMDIRGLTQ GDTATYFCAR QKFYTGGQGW YFDLWGRGTL IVVSSASTKG PSVFPLAPSS KSTSGGTAAL GCLVKDYFPE PVTVSWNSGA LTSGVHTFPA VLQSSGLYSL SSVVTVPSSS LGTQTYICNV NHKPSNTKVD KKVEPKSC |
-分子 #2: PGV04 Fab LC
| 分子 | 名称: PGV04 Fab LC / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: EIVLTQSPGT LSLSPGETAS LSCTAASYGH MTWYQKKPGQ PPKLLIFATS KRASGIPDRF SGSQFGKQYT LTITRMEPED FARYYCQQLE FFGQGTRLEI RRTVAAPSVF IFPPSDEQLK SGTASVVCLL NNFYPREAKV QWKVDNALQS GNSQESVTEQ DSKDSTYSLS ...文字列: EIVLTQSPGT LSLSPGETAS LSCTAASYGH MTWYQKKPGQ PPKLLIFATS KRASGIPDRF SGSQFGKQYT LTITRMEPED FARYYCQQLE FFGQGTRLEI RRTVAAPSVF IFPPSDEQLK SGTASVVCLL NNFYPREAKV QWKVDNALQS GNSQESVTEQ DSKDSTYSLS STLTLSKADY EKHKVYACEV THQGLSSPVT KSFNRGEC |
-分子 #3: AMC011v.4.2_SOSIP
| 分子 | 名称: AMC011v.4.2_SOSIP / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDAMKRGLCC VLLLCGAVFV SPSQEIHARF RRGARAEQLW VTVYYGVPVW KEATTTLFCA SDARAYDTEV RNVWATHACV PTDPNPQEVV LENVTENFNM WKNNMVEQMH EDIISLWDQS LKPCVKLTPL CVTLNCTDLR NATNTNATNT TSSSRGTMEG GEIKNCSFNI ...文字列: MDAMKRGLCC VLLLCGAVFV SPSQEIHARF RRGARAEQLW VTVYYGVPVW KEATTTLFCA SDARAYDTEV RNVWATHACV PTDPNPQEVV LENVTENFNM WKNNMVEQMH EDIISLWDQS LKPCVKLTPL CVTLNCTDLR NATNTNATNT TSSSRGTMEG GEIKNCSFNI TTSMRDKVQK EYALFYKLDV VPIKNDNTSY RLISCNTSVI TQACPKVSFE PIPIHYCAPA GFAILKCNDK KFNGTGPCTN VSTVQCTHGI RPVVSTQLLL NGSLAEEEVV IRSANFTDNA KIIIVQLNKS VEINCTRPNN NTRKSIHIGP GRWFYTTGEI IGDIRQAHCN ISGTKWNDTL KQIVVKLKEQ FGNKTIVFNH SSGGDPEIVM HSFNCGGEFF YCNSTQLFNS TWNDGSNYTG TIVLPCRIKQ IVNMWQEVGK AMYAPPIKGQ IRCSSNITGL ILIRDGGKNR SENTEIFRPG GGDMRDNWRS ELYKYKVVKI EPLGIAPTKC KRRVVQRRRR RRAVGIGAVF LGFLGAAGST MGAASMTLTV QARQLLSGIV QQQNNLLRAP EAQQHLLKLT VWGIKQLQAR VLAVERYLKD QQLLGIWGCS GKLICCTAVP WNTSWSNKSY NQIWNNMTWM EWEREIDNYT SLIYTLIEDS QNQQEKNEQE LLELD |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.71 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 構成要素 - 式: C4H11NO3 / 構成要素 - 名称: Tris 詳細: DDM was added to a concentration of 0.3 mM immediately before freezing. |
| グリッド | モデル: Electron Microscopy Sciences, C-flat / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 雰囲気: OTHER |
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 77.0 K / 最高: 77.0 K |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サンプリング間隔: 5.0 µm / デジタル化 - 画像ごとのフレーム数: 1-35 / 撮影したグリッド数: 1 / 実像数: 3480 / 平均露光時間: 7.0 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 当てはまり具合の基準: MolProbity/RMSD |
|---|
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)