+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8066 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | HKU1 spike with attached foldon domain and wild-type furin-cleavage site | |||||||||
 マップデータ マップデータ | None | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Human coronavirus HKU1 (isolate N5) (ウイルス) Human coronavirus HKU1 (isolate N5) (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 20.0 Å | |||||||||
 データ登録者 データ登録者 | Kirchdoerfer RN / Cottrell CA / Wang N / Pallesen J / Yassine HM / Turner HL / Corbett KS / Graham BS / McLellan JS / Ward AB | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2016 ジャーナル: Nature / 年: 2016タイトル: Pre-fusion structure of a human coronavirus spike protein. 著者: Robert N Kirchdoerfer / Christopher A Cottrell / Nianshuang Wang / Jesper Pallesen / Hadi M Yassine / Hannah L Turner / Kizzmekia S Corbett / Barney S Graham / Jason S McLellan / Andrew B Ward /  要旨: HKU1 is a human betacoronavirus that causes mild yet prevalent respiratory disease, and is related to the zoonotic SARS and MERS betacoronaviruses, which have high fatality rates and pandemic ...HKU1 is a human betacoronavirus that causes mild yet prevalent respiratory disease, and is related to the zoonotic SARS and MERS betacoronaviruses, which have high fatality rates and pandemic potential. Cell tropism and host range is determined in part by the coronavirus spike (S) protein, which binds cellular receptors and mediates membrane fusion. As the largest known class I fusion protein, its size and extensive glycosylation have hindered structural studies of the full ectodomain, thus preventing a molecular understanding of its function and limiting development of effective interventions. Here we present the 4.0 Å resolution structure of the trimeric HKU1 S protein determined using single-particle cryo-electron microscopy. In the pre-fusion conformation, the receptor-binding subunits, S1, rest above the fusion-mediating subunits, S2, preventing their conformational rearrangement. Surprisingly, the S1 C-terminal domains are interdigitated and form extensive quaternary interactions that occlude surfaces known in other coronaviruses to bind protein receptors. These features, along with the location of the two protease sites known to be important for coronavirus entry, provide a structural basis to support a model of membrane fusion mediated by progressive S protein destabilization through receptor binding and proteolytic cleavage. These studies should also serve as a foundation for the structure-based design of betacoronavirus vaccine immunogens. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8066.map.gz emd_8066.map.gz | 16.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8066-v30.xml emd-8066-v30.xml emd-8066.xml emd-8066.xml | 14 KB 14 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8066.png emd_8066.png | 53.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8066 http://ftp.pdbj.org/pub/emdb/structures/EMD-8066 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8066 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8066 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8066_validation.pdf.gz emd_8066_validation.pdf.gz | 78.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8066_full_validation.pdf.gz emd_8066_full_validation.pdf.gz | 77.8 KB | 表示 | |
| XML形式データ |  emd_8066_validation.xml.gz emd_8066_validation.xml.gz | 493 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8066 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8066 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8066 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8066 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8066.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8066.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.05 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
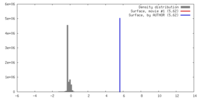
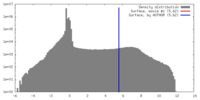
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : HKU1 spike with attached foldon domain and wild-type furin-cleava...
| 全体 | 名称: HKU1 spike with attached foldon domain and wild-type furin-cleavage site |
|---|---|
| 要素 |
|
-超分子 #1: HKU1 spike with attached foldon domain and wild-type furin-cleava...
| 超分子 | 名称: HKU1 spike with attached foldon domain and wild-type furin-cleavage site タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Human coronavirus HKU1 (isolate N5) (ウイルス) Human coronavirus HKU1 (isolate N5) (ウイルス) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: FreeStyle 293F / 組換プラスミド: pVRC8400 Homo sapiens (ヒト) / 組換細胞: FreeStyle 293F / 組換プラスミド: pVRC8400 |
| 分子量 | 理論値: 420 KDa |
-分子 #1: HKU1 spike with attached foldon domain and wild-type furin-cleava...
| 分子 | 名称: HKU1 spike with attached foldon domain and wild-type furin-cleavage site タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human coronavirus HKU1 (isolate N5) (ウイルス) Human coronavirus HKU1 (isolate N5) (ウイルス) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: VIGDFNCTNS FINDYNKTIP RISEDVVDVS LGLGTYYVLN RVYLNTTLLF TGYFPKSGAN FRDLALKGSI YLSTLWYKPP FLSDFNNGIF SKVKNTKLYV NNTLYSEFST IVIGSVFVNT SYTIVVQPHN GILEITACQY TMCEYPHTVC KSKGSIRNES WHIDSSEPLC ...文字列: VIGDFNCTNS FINDYNKTIP RISEDVVDVS LGLGTYYVLN RVYLNTTLLF TGYFPKSGAN FRDLALKGSI YLSTLWYKPP FLSDFNNGIF SKVKNTKLYV NNTLYSEFST IVIGSVFVNT SYTIVVQPHN GILEITACQY TMCEYPHTVC KSKGSIRNES WHIDSSEPLC LFKKNFTYNV SADWLYFHFY QERGVFYAYY ADVGMPTTFL FSLYLGTILS HYYVMPLTCN AISSNTDNET LEYWVTPLSR RQYLLNFDEH GVITNAVDCS SSFLSEIQCK TQSFAPNTGV YDLSGFTVKP VATVYRRIPN LPDCDIDNWL NNVSVPSPLN WERRIFSNCN FNLSTLLRLV HVDSFSCNNL DKSKIFGSCF NSITVDKFAI PNRRRDDLQL GSSGFLQSSN YKIDISSSSC QLYYSLPLVN VTINNFNPSS WNRRYGFGSF NLSSYDVVYS DHCFSVNSDF CPCADPSVVN SCAKSKPPSA ICPAGTKYRH CDLDTTLYVK NWCRCSCLPD PISTYSPNTC PQKKVVVGIG EHCPGLGINE EKCGTQLNHS SCFCSPDAFL GWSFDSCISN NRCNIFSNFI FNGINSGTTC SNDLLYSNTE ISTGVCVNYD LYGITGQGIF KEVSAAYYNN WQNLLYDSNG NIIGFKDFLT NKTYTILPCY SGRVSAAFYQ NSSSPALLYR NLKCSYVLNN ISFISQPFYF DSYLGCVLNA VNLTSYSVSS CDLRMGSGFC IDYALPSSRR KRRGISSPYR FVTFEPFNVS FVNDSVETVG GLFEIQIPTN FTIAGHEEFI QTSSPKVTID CSAFVCSNYA ACHDLLSEYG TFCDNINSIL NEVNDLLDIT QLQVANALMQ GVTLSSNLNT NLHSDVDNID FKSLLGCLGS QCGSSSRSLL EDLLFNKVKL SDVGFVEAYN NCTGGSEIRD LLCVQSFNGI KVLPPILSET QISGYTTAAT VAAMFPPWSA AAGVPFSLNV QYRINGLGVT MDVLNKNQKL IANAFNKALL SIQNGFTATN SALAKIQSVV NANAQALNSL LQQLFNKFGA ISSSLQEILS RLDNLEAQVQ IDRLINGRLT ALNAYVSQQL SDITLIKAGA SRAIEKVNEC VKSQSPRINF CGNGNHILSL VQNAPYGLLF IHFSYKPTSF KTVLVSPGLC LSGDRGIAPK QGYFIKQNDS WMFTGSSYYY PEPISDKNVV FMNSCSVNFT KAPFIYLNNS IPNLSDFEAE LSLWFKNHTS IAPGSGYIPE APRDGQAYVR KDGEWVLLST FLGLEVLFQ |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.25 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||
| 染色 | タイプ: NEGATIVE / 材質: uranyl formate 詳細: 3uL sample was applied to grid for 30 seconds and then blotted. Grids were stained with 3uL 1% uranyl formate for 60 seconds followed by blotting. | |||||||||
| グリッド | 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI SPIRIT |
|---|---|
| 撮影 | フィルム・検出器のモデル: TVIPS TEMCAM-F416 (4k x 4k) 撮影したグリッド数: 1 / 実像数: 123 / 平均露光時間: 1.0 sec. / 平均電子線量: 28.0 e/Å2 詳細: Images were collected using Legionon and processed using Appion. |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 52000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
| 実験機器 |  モデル: Tecnai Spirit / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)